哈喽哈喽,你的小可爱小花突然出现~今天和大家聊一聊NMF,“非负矩阵分解”,NMF方法在生信领域非常关键,可以用于降维、异常值检测、特征提取等等很多方面,在进行实操之前,小花先给大家讲讲NMF非负矩阵分解的原理!
首先想象一下你正在玩拼图游戏。你有一个巨大的、充满各种形状和颜色的拼图,这就是你的非负矩阵V。现在,你想要将这个巨大的拼图分成两部分,每一部分都是由一些小拼图块组成的,这些小拼图块可以组合在一起还原成原始拼图。
我们把这两部分放到两个特殊的盒子里,一个叫W盒子,而另一个叫H盒子。这两个盒子的特殊之处在于,里面的拼图块都是非负的,没有负号或黑暗的拼图块,说到这里,就和NM这部分单词对应上啦,而生物数据很多都是非负的,如果分解出负值就缺乏了生物学意义,这就使得NMF相比奇异值分解(SVD)等方法来说更有优势。
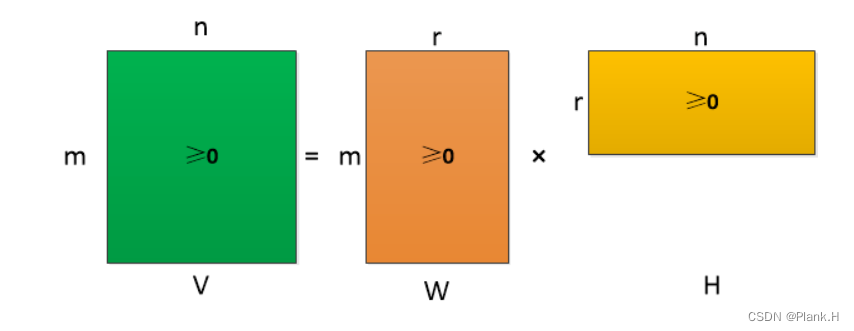
在我们拼图的过程中,你可以用W和H来代替原始的拼图,实现了数据的降维,同时减少了存储空间的需求。这就好像是将一个庞大的拼图,变成了一个小而精致的拼图,更容易管理和展示。
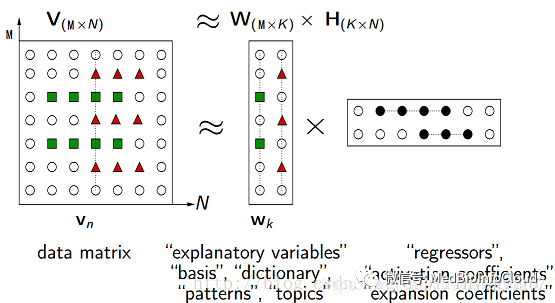
上面这张图更容易理解,假设原先是M×N的基因样本矩阵,行为基因,列为样本,之后将其分为了W矩阵和H矩阵,如果用H来表示整个矩阵降维后的结果,便是每个细胞用一个二维向量来表示,这样就实现了降维,从原先的7维降到了2维,比如之前在Seurat包中进行PCA降维,就可以将RunPCA中的参数pca改为nmf,具体可以参考下面这段代码,将原始数据进行矩阵分解后就可以聚类,如果还是有小伙伴不懂,可以看看小花之前讲的Seurat包,把这段代码插进去就好啦。
vm <- pbmc@assays$RNA@scale.data
res <- nmf(vm, 10, method = “snmf”, seed = ‘nndsvd’)
## 分解结果返回suerat对象
pbmc@reductions$nmf <- pbmc@reductions$pca
pbmc@reductions$nmf@cell.embeddings <- t(coef(res))
pbmc@reductions$nmf@feature.loadings <- basis(res)
## 使用nmf的分解结果降维聚类
set.seed(219)
pbmc.nmf <- RunUMAP(pbmc, reduction = ‘nmf’, dims = 1:10) %>%
NMF除去降维外,也可以用于聚类,不过这里的聚类指的是使用降维后得到的特征综合已知聚类方法(如K-meas)进行聚类,下面给大家演示一下如何用R包nmf对一个表达矩阵进行聚类,或者说对肿瘤进行分型:
NMF这个R包的安装很简单,只需要install.packages(‘NMF’)就可以啦,给大家讲讲这个函数的参数
nmf(x, rank,
method, seed = nmf.getOption(“default.seed”),
rng = NULL, nrun = if (length(rank) > 1) 30 else 1,
model = NULL, .options = list(),
.pbackend = nmf.getOption(“pbackend”),
.callback = NULL, …)
- x:这里就是我们的表达矩阵;
- rank:简单理解是就是我们想分成几个cluster,可以是一个固定的数值,也可以是一个范围(例如2:7)
- method:就是NMF算法的选择,总共有11种,可通过nmfAlgorithm()函数查看,默认brunet
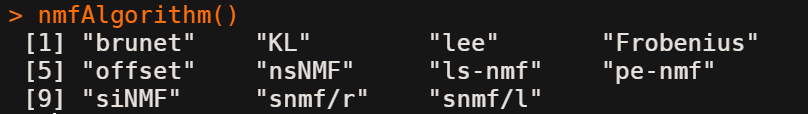
- nrun:范围内每个值的迭代次数;
- options:可以设置是否保留每次的运算结果:keep.all=T。例:.options=list(keep.all=TRUE)
在给大家说明了我们在分析中用到的R包后就要开始正式书写啦,我们前面已经提到这里我们举的是NMF进行聚类的例子,说到聚类,必不可少的一个步骤便是确定到底要分成几类,在这里我们通过其中的rank参数进行确定,下面就是我们的代码啦:
n <- 50;
counts <- c(5, 5, 8);
V <- syntheticNMF(n, counts)
我们在这里生成一个50×18的随机矩阵,然后进行下面的NMF分析:
estimate <- nmf(V, rank=2:10, method=”brunet”, nrun=5, seed=123)
这行代码是全文的核心!参数在上面已经解释过啦,这里就不过多阐述了
plot(estimate)
consensusmap(estimate)
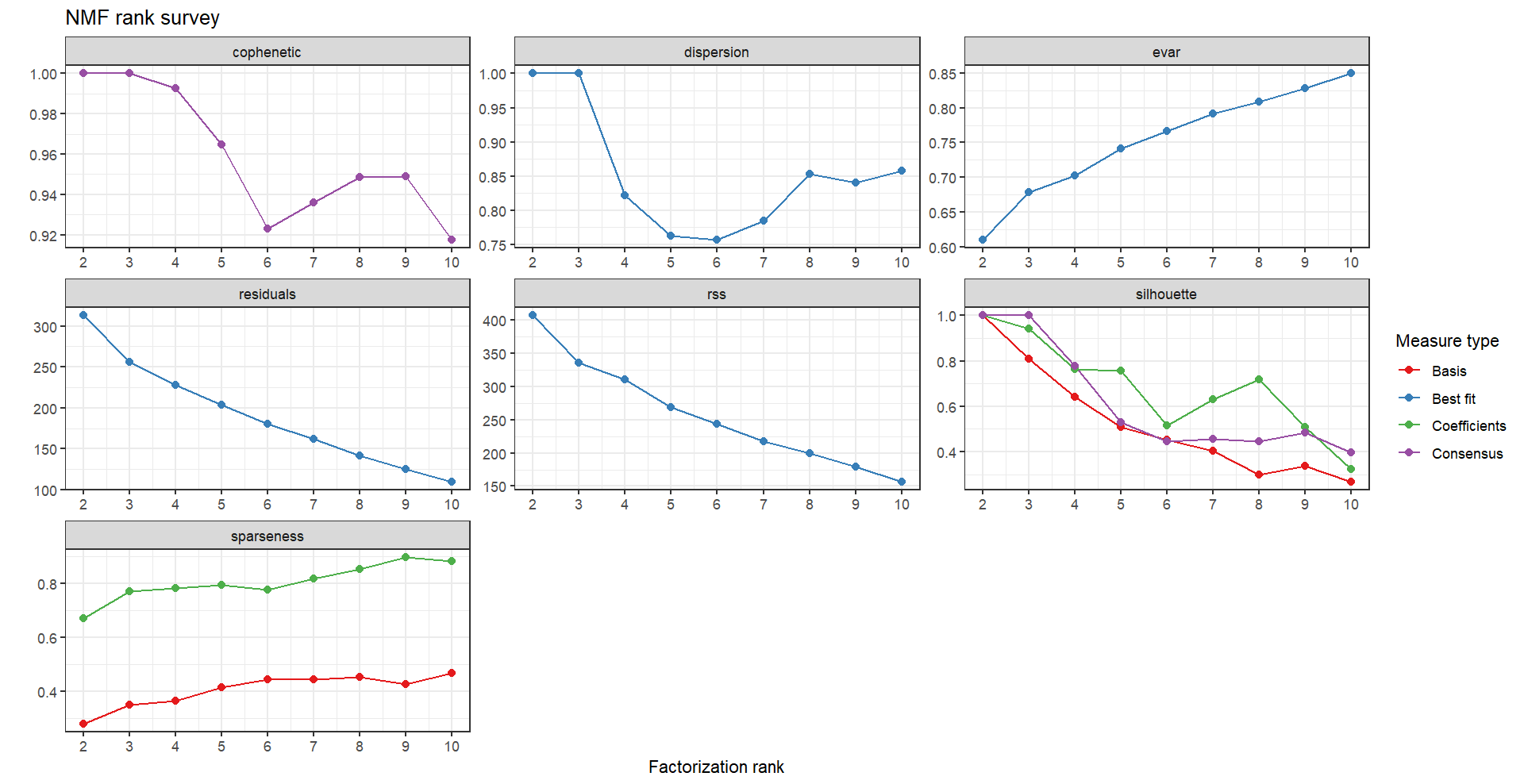
判断最佳rank值的准则就是,cophenetic值随K变化的最大变动的前点,上面结果中cophenetic值在rank为5-6时是第一个变化最大的拐点,所以选择最佳rank值为5。
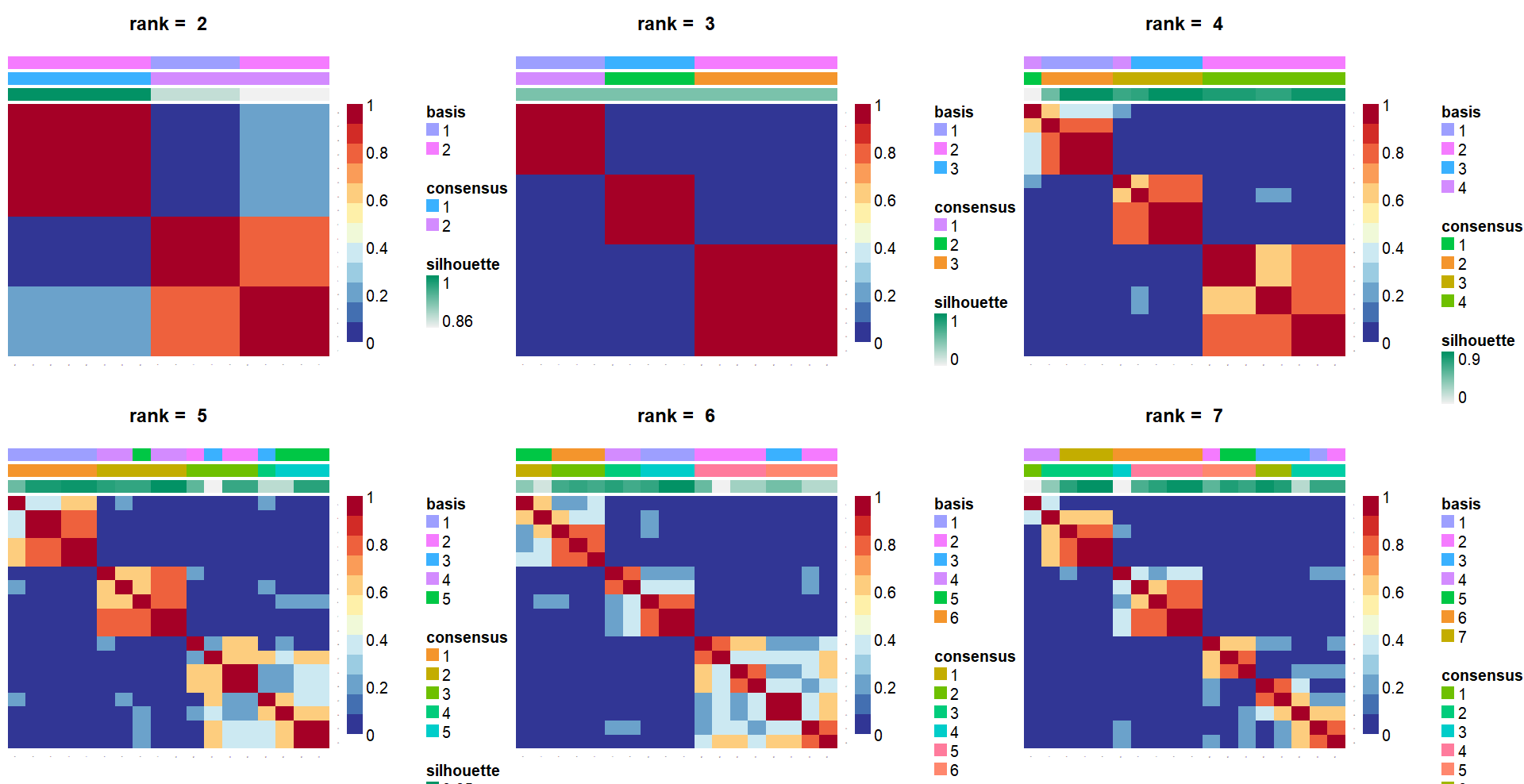
estimate.rank5 <- nmf(V,5,nrun=10,seed=555555,method = ‘brunet’)
group <- predict(estimate.rank5)
group <- as.data.frame(group)
当确定了rank5为最好的聚类结果时,可以重新使用nmf进行聚类,最后在group中查看对应的分型。
最后是总结环节啦,非负矩阵分解 (NMF) 是一种无监督学习技术,可以将一个非负矩阵分解为两个非负矩阵的乘积,从而提取出数据的内在结构和特征。NMF有很多应用:
- 基因表达数据分析:NMF 可以用于从基因表达数据中识别出不同的基因表达模式,反映出细胞的功能和状态。NMF 还可以用于对细胞进行聚类和分类,发现细胞的异质性和亚群。
- 图像处理和识别:NMF 可以用于从图像数据中提取出局部特征,实现图像的降维和压缩。NMF 还可以用于图像的分割和重构,以及人脸识别等应用。
- 文本挖掘和主题建模:NMF 可以用于从文本数据中提取出关键词和主题,实现文本的表示和分类。NMF 还可以用于文本的相似度计算和聚类,以及信息检索和推荐等。
其他用处有待大家挖掘,比如小花见过使用NMF从cfDNA测序数据中提取出有生物意义的一些片段特征进行预测,对于细胞分群效果不理想,如上皮细胞存在异质性等,可以考虑使用非负矩阵分解(NMF)的方式去定义元程序(meta program,即功能模块)和元标签(meta label,即功能基因集)。
感谢大家看到这里!我们的NMF之旅就结束啦,如果有问题可以给小花留言哦,最后再来宣传宣传云生信- 学生物信息学(http://www.biocloudservice.com/home.html)——聚类降维等很多功能一体化集成的网站!欢迎大家多多捧场~