名称:基于表达信息挖掘与靶基因相关的基因
网址:http://www.biocloudservice.com/123/123.php
功能: 在生信分析中,我们往往希望对我们关注的某一个基因进行下游功能的探索,然而,单个基因想要研究,大多数情况下只能是通过实验进行,通常会对该基因进行敲除或过表达后进行测序,从而和正常组进行比较,得到差异基因认为是受该基因影响的,但是这样做一是耗时耗力,而是得到的差异基因仍然是一堆,我们仍然不知道哪些基因和该基因密切相关。皮尔森相关系数和斯皮尔曼相关系数为研究基因之间的表达相关性提供了很好的思路,因此本软件通过基于基因在各个样本中的表达矩阵,利用皮尔森相关系数和斯皮尔曼相关系数计算原理,分别获得各个基因与关注基因的相关性排名,通过给定的基因个数N,绘制相关性最强的TOPN基因和关注基因的散点分布图,并标识出具体的相关系数和显著性p.value值,从而为下游功能研究提供参考。
操作:
- 对参数进行设置。这里,必须输入参数有3个,分别是-Eset,表示基因表达矩阵文件,以基因为行,样本为列,保存为csv文件;-symbol,表示关注的基因名称,为gene symbol格式,必须包含在基因表达矩阵的行名中;-topnumber,表示预期绘制的与关注基因最相关的基因个数,这里假如是6,则会绘制与关注基因最正相关的6个和最负相关的6个基因的相关性散点图。
- 点击上传数据 输入***文件,输入的文件名必须和示例文件名一致,且都是英文无特殊字符。 如果使用自有数据时,需注意文件名和对应的内容要匹配,以及输入数据里的行名列名等等。
- 如有非文件类参数,点击 输入参数对应的值,默认比如 pvalue值是0.05 gene 值是 TP53
操作截图:
1.示例数据运行操作截图:
点击 运行示例数据,然后等待弹出的网页运行完成,具体耗时根据程序来定,一般2-3分钟内完成。
- 运行自有数据操作截图
点击 上传数据–上传–上传需要的文件(可以参考输入数据模板,可能1个或多个)
点击 运行自有数据,等待程序运行结束后新弹出的网页就会显示结果,拉到最下方,点击“结果下载”或“下载全部文件”就可以批量下载了。
输入文件名:
输入文件名1:expr.csv
输入其他参数
Symbol:HDAC1
Topnumber:6
输入文件界面展示截图示例
基因表达矩阵输入文件(基因为行,样本为列)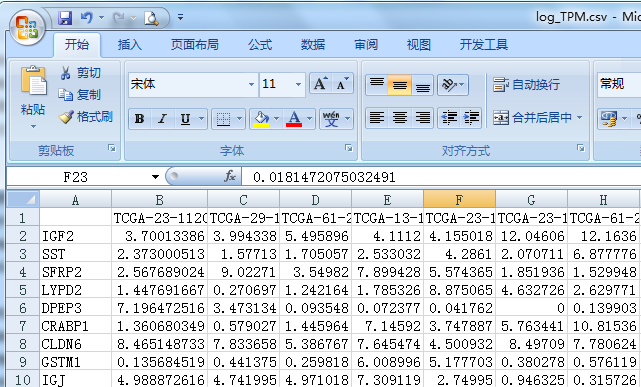
输出文件:
共输出一个表格csv格式文件,一个pdf格式的图片文件
1.HDAC1_cor.pdf
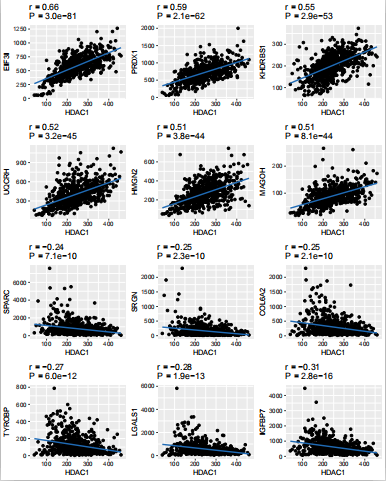
该图表示各个基因与关注基因的相关性散点图,纵轴名称是其它各个基因symbol,横轴名称为关注基因symbol,最上方P值表示显著行p.value值,r值表示pearson相关系数
- HDAC1other_gene_cor.csv
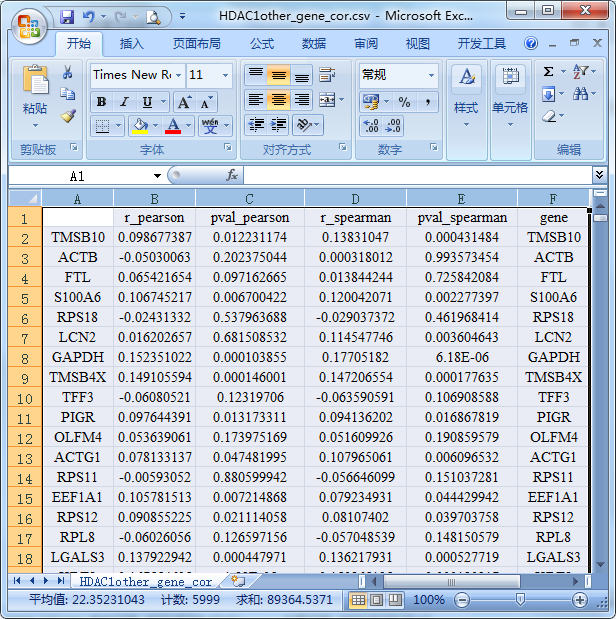
该表格表示各个基因和关注基因之间的相关系数及p.value值,第二和第三列分别表示计算得到的各个基因和关注基因的pearson相关系数和对应的显著性p.value值,第四和第五列分别表示计算得到的各个基因和关注基因的spearman相关系数和对应的显著性p.value值,第六列为对应的基因symbol
服务器运行流程
1、软件内容及流程:
共包含1个R语言脚本,脚本介绍如下:
(1)batchCorrelation.r
程序名称:
batchCorrelation.r
功能描述:
在生信分析中,我们往往希望对我们关注的某一个基因进行下游功能的探索,然而,单个基因想要研究,大多数情况下只能是通过实验进行,通常会对该基因进行敲除或过表达后进行测序,从而和正常组进行比较,得到差异基因认为是受该基因影响的,但是这样做一是耗时耗力,而是得到的差异基因仍然是一堆,我们仍然不知道哪些基因和该基因密切相关。皮尔森相关系数和斯皮尔曼相关系数为研究基因之间的表达相关性提供了很好的思路,因此本软件通过基于基因在各个样本中的表达矩阵,利用皮尔森相关系数和斯皮尔曼相关系数计算原理,分别获得各个基因与关注基因的相关性排名,通过给定的基因个数N,绘制相关性最强的TOPN基因和关注基因的散点分布图,并标识出具体的相关系数和显著性p.value值,从而为下游功能研究提供参考。
使用方法:
batchCorrelation.r -Eset= -symbol= -topnumber=
参数说明:
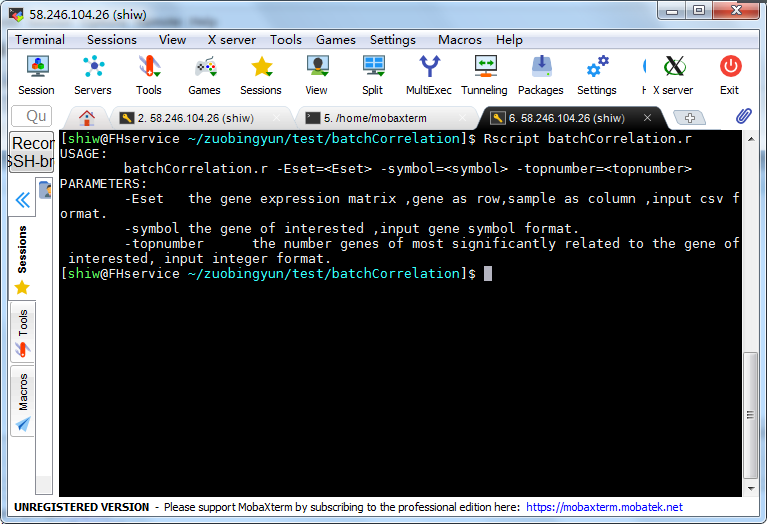
USAGE:
batchCorrelation.r -Eset=<Eset> -symbol=<symbol> -topnumber=<topnumber>
PARAMETERS:
-Eset the gene expression matrix ,gene as row,sample as column, input csv format.
-symbol the gene of interested ,input gene symbol format.
-topnumber the number genes of most significantly related to the gene of interested, input integer format.
Rscript /var/www/r/batchCorrelation.r -Eset=’expr.csv’ -symbol=”HDAC1″ -topnumber=6
操作步骤:
1、打开命令行界面,输入“Rscript batchCorrelation.r”调阅帮助文档,确定该程序所需的输入文件。
2、用户根据帮助文档中的参数说明内容,对参数进行设置。这里,必须输入参数有3个,分别是-Eset,表示基因表达矩阵文件,以基因为行,样本为列,保存为csv文件;-symbol,表示关注的基因名称,为gene symbol格式,必须包含在基因表达矩阵的行名中;-topnumber,表示预期绘制的与关注基因最相关的基因个数,这里假如是6,则会绘制与关注基因最正相关的6个和最负相关的6个基因的相关性散点图。
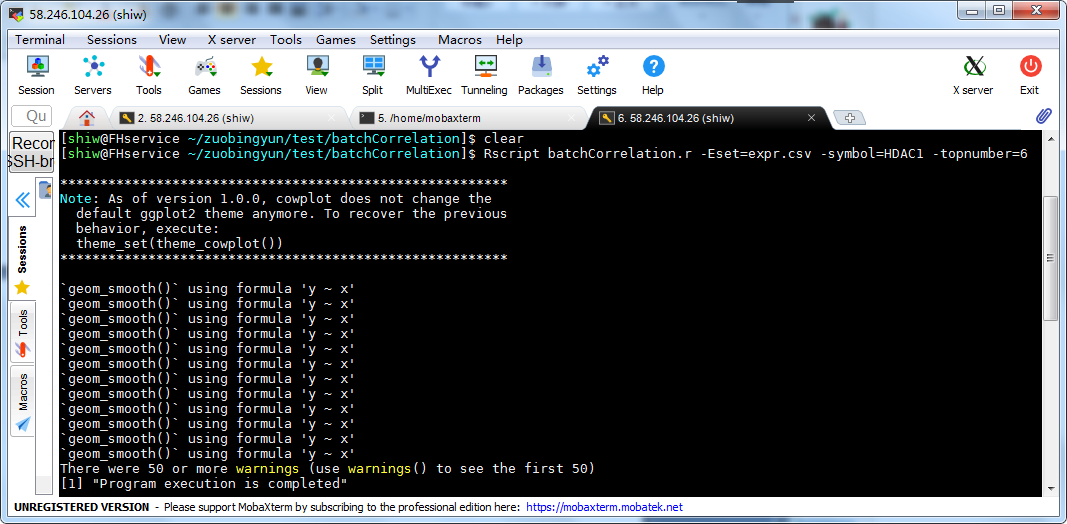
3、完成参数提交后,按下回车键,整个程序即正式开始进入执行。每步执行内容都会给出提示。程序执行完毕后,界面会显示”Program execution is completed”结束语。
操作界面截图:
- 参数说明文档调阅截图说明
- 输入文件界面展示截图示例
基因表达矩阵输入文件(基因为行,样本为列)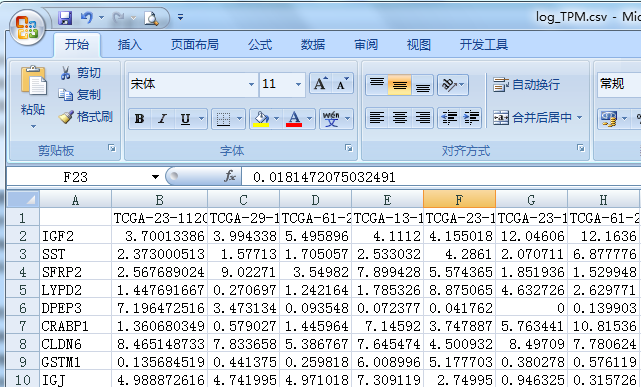
- 程序运行界面截图
程序运行完毕提示:
- 程序运行结果界面展示
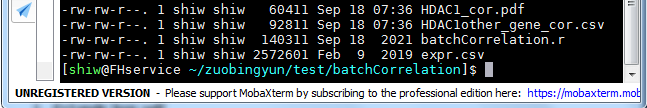
输出文件:
共输出一个表格csv格式文件,一个pdf格式的图片文件
1.HDAC1_cor.pdf
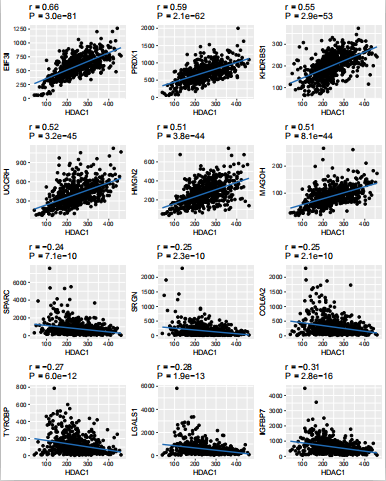
该图表示各个基因与关注基因的相关性散点图,纵轴名称是其它各个基因symbol,横轴名称为关注基因symbol,最上方P值表示显著行p.value值,r值表示pearson相关系数
- HDAC1other_gene_cor.csv
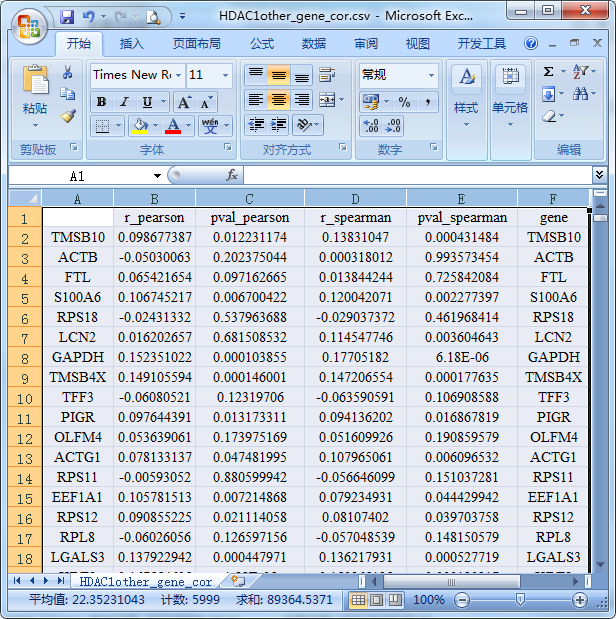
该表格表示各个基因和关注基因之间的相关系数及p.value值,第二和第三列分别表示计算得到的各个基因和关注基因的pearson相关系数和对应的显著性p.value值,第四和第五列分别表示计算得到的各个基因和关注基因的spearman相关系数和对应的显著性p.value值,第六列为对应的基因symbol
2、流程图
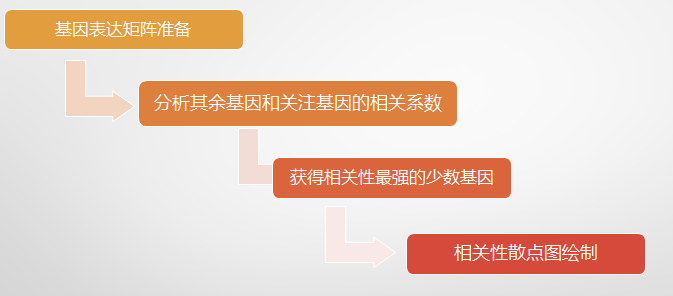
3、创新点:
本软件通过基于基因在各个样本中的表达矩阵,利用皮尔森相关系数和斯皮尔曼相关系数计算原理,分别获得各个基因与关注基因的相关性排名,通过给定的基因个数N,绘制相关性最强的TOPN基因和关注基因的散点分布图,并标识出具体的相关系数和显著性p.value值。
设置参数少,用户只需要输入基因在所有样本中的表达值矩阵,并设置好感兴趣或关注的基因symbol与预期得到的相关性最强的基因个数,软件将自行计算出矩阵中其它各个基因和关注基因之间的皮尔森相关系数和斯皮尔曼相关系数,并绘制与关注基因相关性最强的少数基因和关注基因的分布散点图。