鲜活的热图,诠释免疫细胞组成:使用CIBERSORT并实现可视化分析!
基于之前对CIBERSORT软件包的介绍和使用,今天小果要教大家如何在使用CIBERSORT包的前提下对其结果进行可视化,可视化我们要用到的R包主要为ggplot2,感兴趣的话就和小果一起来看一下吧!
准备工作及免疫细胞分析
这一部分其实在之前小果已经给大家做过详细的介绍,在这里我们再次简单的回顾一下吧!这一步我们主要要做的工作有:
- 安装以下 R 库:ggplot2, ggpubr, reshape2, dplyr, CIBERSORT;
- 下载 LM22.txt数据集 和 TCGA deg_fpkm.tsv 数据文件;
- 从 LM22.txt 和 TCGA deg_fpkm.tsv 文件中读取基因表达数据;
- 使用 CIBERSORT 库对基因表达数据进行免疫细胞类型的分析,并使用 CIBERSORT 函数计算出各种免疫细胞类型的比例;
- 将 CIBERSORT 计算结果输出到文件中
library(ggplot2)
library(reshape2)
library(ggpubr)
library(dplyr)
lm22.file <- “LM22.txt”
rawdata_file.file<-“TCGA deg_fpkm.tsv”
cibersort_file <- “TCGA cibersort.txt”
library(‘CIBERSORT’)
#计算免疫细胞
TCGA_TME.results <- CIBERSORT(lm22.file ,rawdata_file.file, perm = 50, QN = F)
TCGA_TME.results
write.csv(TCGA_TME.results, “TCGA_CIBERSORT_Results_fromRcode.csv”)
现在,你已经成功完成了免疫细胞类型分析哦!怎么样,是不是很简单呢!
将CIBERSORT的输出数据进行整理,以适应ggplot2的绘图要求
data <- t(TCGA_TME.results[,-c(9:11)])
col=rainbow(nrow(data),s=0.85,v=0.85)
df.long <- reshape2::melt(data, value.name=’abundance’, variable.name=’group’)
使用ggplot2包生成热图
ggplot(df.long, aes(x=Var2, y=abundance, group=Var1)) +
theme_classic()+
geom_bar(stat=”identity”, position=”fill”, aes(fill=Var1))+
scale_y_continuous(labels = scales::percent) +
labs(x=”,y=’Relative Percent’,title=’Immune Cell Subset Proportions’)+
scale_fill_manual(values =col)+
theme( panel.grid=element_blank(),legend.title=element_blank(),
plot.title = element_text(hjust = 0.5),
axis.text.x = element_text(angle =90))
生成的结果如下图所示: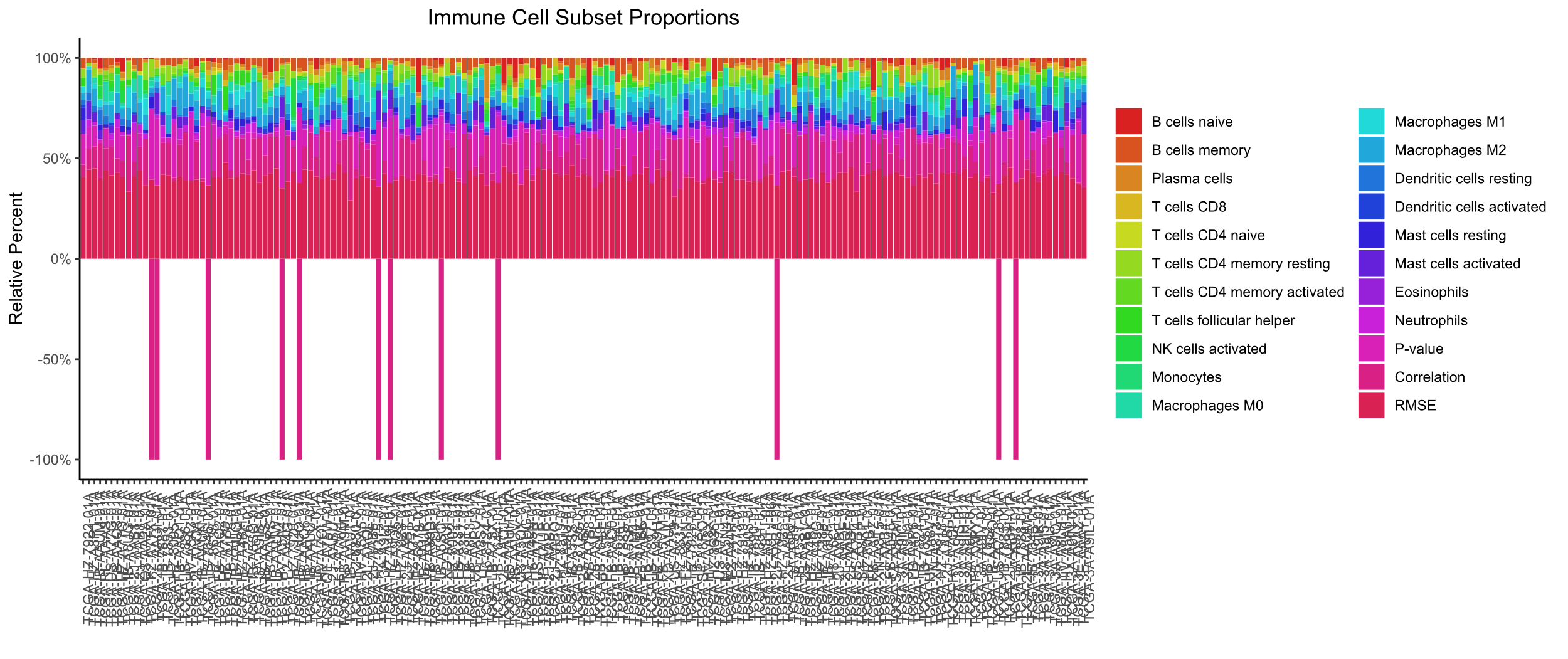
将图像保存为PDF格式
ggsave(“cibersort_barplot.pdf”,height=10,width=25)
在上述代码中,我们使用reshape2包的melt函数将CIBERSORT的输出数据进行整理,生成长格式的数据框。然后,我们使用ggplot2包的geom_bar函数生成堆积条形图,并使用scale_fill_manual函数设置颜色,theme函数设置样式,labs函数设置图像标题和轴标签。最后,我们使用ggsave函数将图像保存为PDF格式。
到此为止,我们已经成功地完成了免疫细胞比例分析,并可视化了结果。我们可以根据分析结果来进一步研究免疫细胞在肿瘤组织中的作用和机制,为肿瘤免疫治疗提供科学依据和指导!怎么样,今天的热图绘制你学会怎么用了吗!
更多学习资源请大家移步小果专属云生信平台搜索更多资源哦!
小果专属云生信平台:云生信 – 学生物信息学 (biocloudservice.com)
云生信平台也有免疫专版的学习模块哦,快来找到你想学习的专属模块吧!