Estimate包计算多种得分并用ggplot实现可视化:从免疫基质得分到彩虹之桥!
嘿大家!好久不见,今天我们来学习一个新的R包,它可以用来进行免疫得分的计算,并绘制箱线图进行结果展示。感兴趣的话就和小果一起学起来吧!
什么是estimate包?
在这之前,小果先给大家对estimate包做一个简单的介绍。
estimate是一个在肿瘤组学研究中常用的R包,其主要功能是用于评估肿瘤组织中免疫细胞浸润的程度,并对免疫相关的信号通路进行分析。estimate基于基因表达数据进行计算,主要利用了免疫和肿瘤细胞特异的基因表达标记,并根据这些标记的表达水平估算组织中免疫和非免疫细胞的数量。最终,estimate会输出三个指标:ImmuneScore、StromalScore和ESTIMATEScore,用于评估免疫和成纤维细胞浸润的程度及其影响。
小果提醒,今天我们要学的就是通过estimate计算ImmuneScore值并将其可视化,同时在学会计算ImmuneScore(免疫得分)的基础上小果还会向大家介绍如何计算StromalScore(基质得分),那么大家就和小果一起来看一下吧!
数据导入及预处理
首先我们需要安装并载入一个叫做“estimate”的包,这个包可以用来计算Immune Score的分值哦。
library(estimate)
接下来我们来导入我们准备好的数据:
rawdata_file <- “tumor_all_gene_exp.txt”
接下来我们使用“filterCommonGenes”函数对数据集进行过滤,并将结果保存到“my.gct”的文件中。在这个函数中,我们还要指定“GeneSymbol”作为基因的ID:
filterCommonGenes(input.f=rawdata_file,
output.f=”my.gct”,
id=”GeneSymbol”)
计算免疫得分
然后我们需要使用“estimateScore”函数来计算免疫得分。在这个函数中,我们需要指定输入文件的路径,以及输出文件的路径。我们还要指定数据的平台(例如affymetrix):
estimateScore(input.ds = “my.gct”,
output.ds=”estimate_score.gct”,
platform=”affymetrix”)
接下来,我们需要读取输出文件“estimate_score.gct”中的数据,并将其保存到名为“raw_data”的数据集中:
raw_data = read.table(“estimate_score.gct”,skip = 2,header = F,quote = ”,sep = ‘\t’,stringsAsFactors=FALSE)
rownames(raw_data)=raw_data[,1]
raw_data= as.data.frame(t(raw_data[,3:ncol(raw_data)]))
row.names(raw_data)<-c(1:nrow(raw_data))
colnames(raw_data)[1] <- c(“sample”)
for(i in 2:5){
raw_data[,i] <- as.numeric(as.character(raw_data[,i]))
}
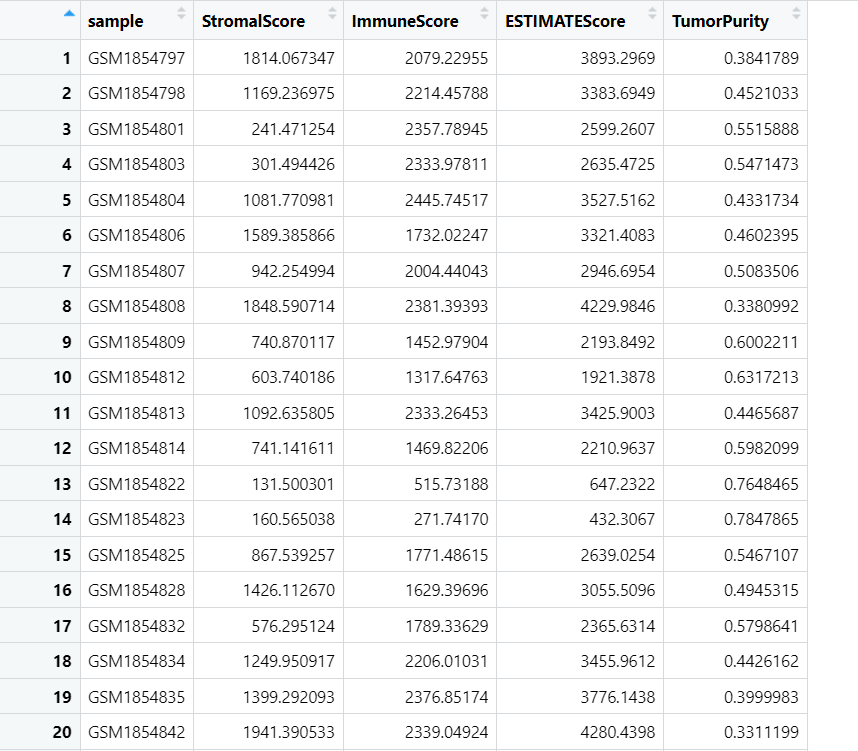 我们一起来看下输出的数据吧:
我们一起来看下输出的数据吧:
然后,我们需要读取准备好的“group.txt”的文件,这个文件包含了每个样本的名称以及其对应的集群,我们需要将其与上面的结果文件合并才能方便之后的可视化工作:
phenotype_file <- read.table(‘group.txt’,header = T,sep = ‘\t’,quote = ”,stringsAsFactors=FALSE)
phenotype_file <- phenotype_file[phenotype_file[,1] %in% raw_data[,”sample”],]
merge_matrix <- left_join(phenotype_file,raw_data,by=”sample”) #合并数据集
my_comparisons <- list( c(“cluster1”, “cluster2”),c(“cluster2″,”cluster3”),c(“cluster1”, “cluster3”))
计算结果可视化
在上面的步骤中,我们成功的计算出了Immune Score的值,现在我们通过我们熟悉的ggplot2包来实现计算结果的可视化:
- 设置my_labely标签
max_labely <- max(merge_matrix[,”ImmuneScore”])
min_labely <- min(merge_matrix[,”ImmuneScore”])
my_labely <- c(max_labely+1350, max_labely+1000, max_labely+650, max_labely+300)
- 绘制箱线图
ggboxplot(merge_matrix, x = “Group”, y = “ImmuneScore”,
color = “black”,alpha = 0.8,size=.3,
add = “jitter”,
add.params = list(color = “Group”,alpha = .5,size = 0.8)) +
stat_compare_means(comparisons = my_comparisons,method = “t.test”,
label.y = my_labely,na.rm=TRUE)+
stat_summary(fun.y = “mean”,
geom = “point”,
color = “#485c70”,
shape = 8,
size = 3) +
- 添加标题和调整绘图细节
ggtitle(“Immune Score”) +theme_bw()+
theme(legend.position=”none”,
plot.title = element_text(size=15,face=”bold”,hjust = 0.5),
axis.text.x=element_text(colour=”black”),
axis.text.y=element_text(colour=”black”),
panel.grid.major = element_blank(),
panel.grid.minor = element_blank()) +
- 设置x轴和y轴名称以及y轴显示范围
ylab(“Immune Score”) +
xlab(“Group”) +
ylim(min_labely-500, max_labely+1500)
box_plot_pdf <- “path/to/immune_score_box.pdf”
ggsave(box_plot_pdf,width=17.8,height=17.8,units = ‘cm’, dpi = 500)
最后,我们使用 ggsave 函数将绘制好的图保存为 PDF 格式。
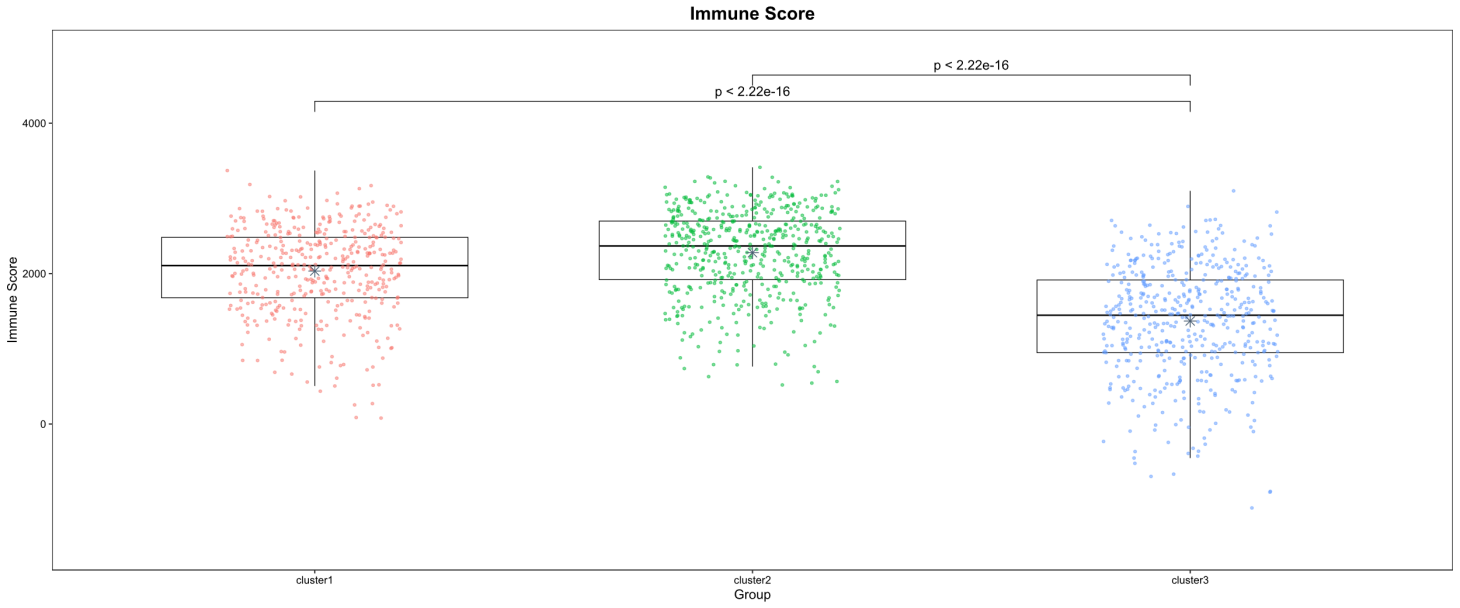 现在和效果一起来看下最后绘制好的图长什么样吧!
现在和效果一起来看下最后绘制好的图长什么样吧!
怎么样,你学会怎么使用estimate包了嘛? 更多学习资源请大家移步小果专属云生信平台搜索更多资源哦!
StromalScore
有了计算ImmuneScore的基础,计算StromalScore也是同样的计算方法和可视化方法,只要提取的是结果文件中的StromalScore列即可:
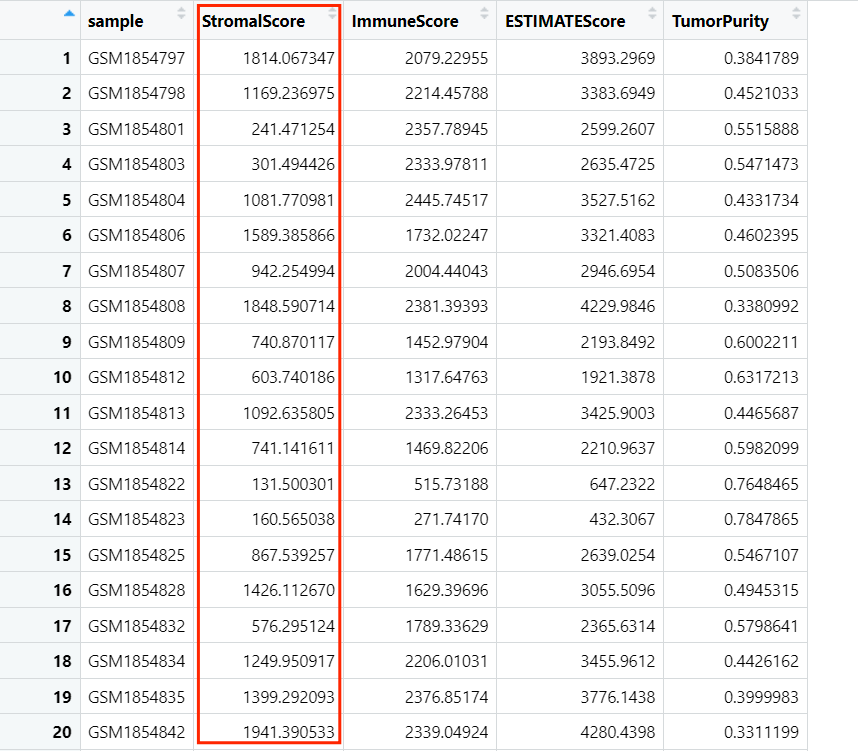
那么我们再来看看最后StromalScore的可视化结果:
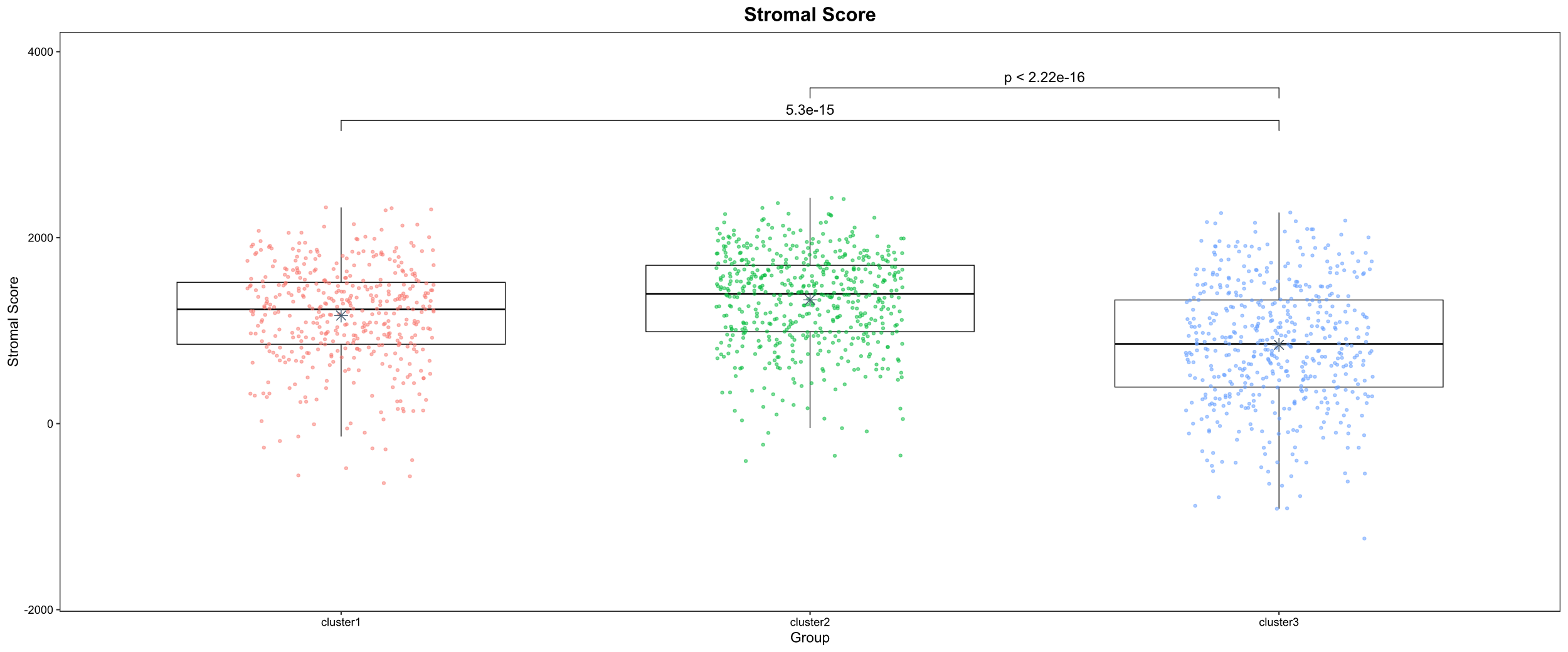
怎么样,今天的图你学会怎么绘制了吗?
小果专属云生信平台:云生信 – 学生物信息学 (biocloudservice.com)
 云生信平台也有免疫专版的学习模块哦,快来找到你想学习的专属模块吧!
云生信平台也有免疫专版的学习模块哦,快来找到你想学习的专属模块吧!