
小云最近阅读了许多的文献,心里一直在思考,科研人员如何发表成功的高分的生信文章。最终呢小云虽然没有完全发掘其中的奥秘,但是小云发现一个规律,这些高分文章中对数据的把控是很到位,如何理解呢,小云看的Nature级别的文章中,他们都数据量都很庞大,
所以小云就想说,小伙伴如果想发高分文章一开始处理的数据一定不能少,样本量够大,这样也可以侧面的说明工作量大,那么要是解决这个问题,后续的思路等就迎刃而解了。

说到数据,小伙伴做分析要是用很大的数据,那就少不了对数据的质控。那该如何指控呢,小云告诉小伙伴用的最多的就是主成分分析(PCA),
目前许多的生信分组虽然他们经常会做PCA分析,但是他们都是很普通的可视化方式,最多的就是横纵坐标的PCA二维图,再者就是三维的PCA图。小云想教给小伙伴不一样方式的PCA图,然后就发现一篇Nature文章中的PCA图,这幅图打破正常我们见过的PCA图,我们先看图在了解一下这个图的特色,这是来自IF影响因子有46.9《Natue biotechnology》中的《Removing unwanted variation from large-scale RNA sequencing data with PRPS》图2a,来看一下:
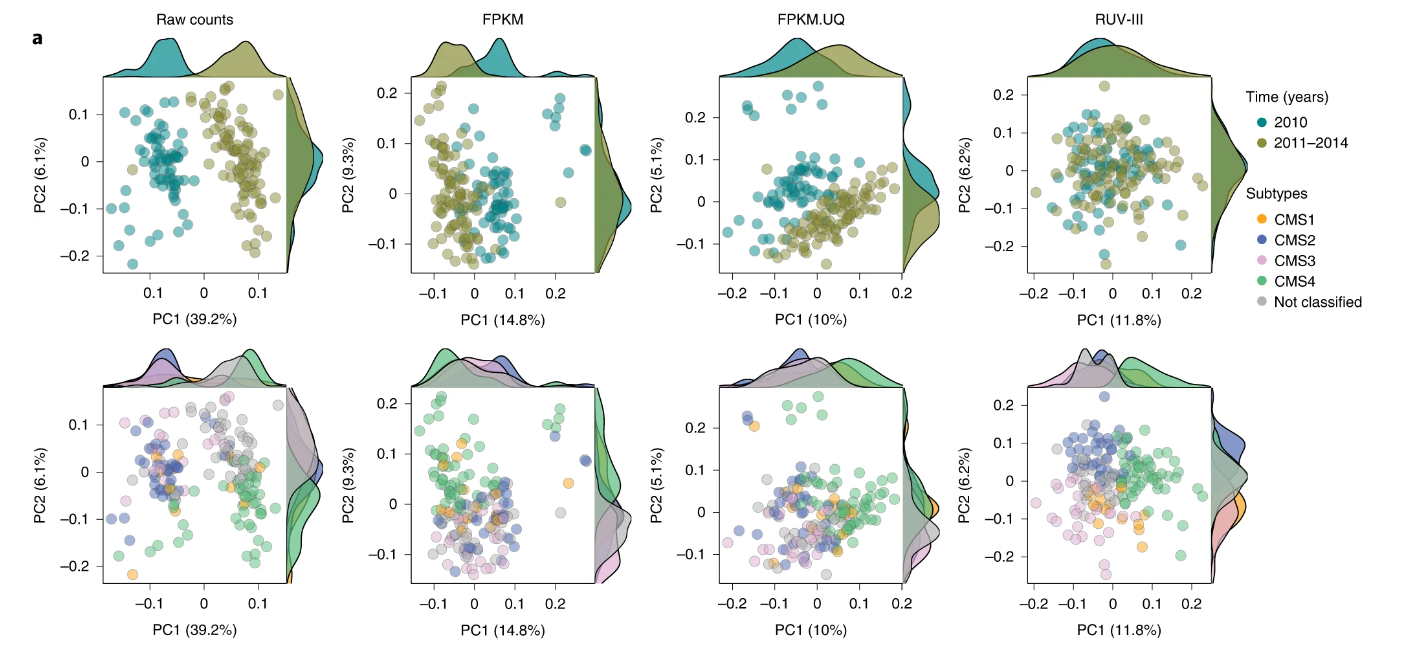
小伙伴看的第一眼是不是觉得这不是PCA图,小云也是第一眼就被吸引了,这幅图,PCA主要的内容应有尽有,并且还非常的好看,那么今天小云就带小伙伴去复现这张图,我们以后做PCA就可以用到这种图了,
作图的前提呢,小云还是带小伙伴了解一下什么是PCA,我们也要明白为什么要做这个图,其实主成分分析属于一种降维算法,当我们的数据量很庞大的时候就需要去把数据降维,而降维呢就是一种对高维度特征数据预处理方法。降维是将高维度的数据保留下最重要的一些特征,去除噪声和不重要的特征,从而实现提升数据处理速度的目的。
在实际的生产和应用中,降维在一定的信息损失范围内,可以为我们节省大量的时间和成本。降维也成为应用非常广泛的数据预处理方法。
这么做是有利于我们分析的,可以使得数据集更易使用。也可以使得结果容易理解。
那么话不多说,我们就开始下面的学习吧!
这里呢小云还要说明一下,我们做PCA,数据库很大的花,那么我们操作占用内存比较大,所以建议各位小伙伴可以使用服务器去运行,如果没有自己的服务器也没有关系,可以欢迎联系我们使用服务器租赁~
好了,我们开始吧!
我们在做PCA分析的时候,因为对许多数据进行处理,这就需要用到一些函数去计算,
这次小云就分享给小伙伴两个函数,来实现我们这次的复现内容,
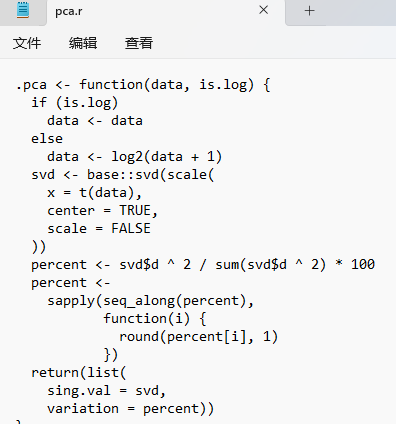
第一个就是我们用来计算主成分分析的pca函数
.pca <- function(data, is.log) {
if (is.log)
data <- data
else
data <- log2(data + 1)
svd <- base::svd(scale(
x = t(data),
center = TRUE,
scale = FALSE
))
percent <- svd$d ^ 2 / sum(svd$d ^ 2) * 100
percent <-
sapply(seq_along(percent),
function(i) {
round(percent[i], 1)
})
return(list(
sing.val = svd,
variation = percent))
}
这个函数可以计算我们的所有的数据,小伙伴也不用理解这个函数具体意思,
第二个函数就是用来作图的,这个就需要用到我们的老朋友ggplot2包中的ggpubr还有cowplot包,
来看一下:
.scatter.density.pc <- function(
pcs,
pc.var,
group.name,
group,
color,
strokeSize,
pointSize,
strokeColor,
alpha,
title
){
pair.pcs <- utils::combn(ncol(pcs), 2)
pList <- list()
for(i in 1:ncol(pair.pcs)){
if(i == 1){
x <- pair.pcs[1,i]
y <- pair.pcs[2,i]
p <- ggplot(mapping = aes(
x = pcs[,x],
y = pcs[,y],
fill = group)) +
xlab(paste0(‘PC’, x, ‘ (‘, pc.var[x], ‘%)’)) +
ylab(paste0(‘PC’, y, ‘ (‘, pc.var[y], ‘%)’)) +
geom_point(
aes(fill = group),
pch = 21,
color = strokeColor,
stroke = strokeSize,
size = pointSize,
alpha = alpha) +
scale_x_continuous(breaks = scales::pretty_breaks(n = 5)) +
scale_y_continuous(breaks = scales::pretty_breaks(n = 5)) +
ggtitle(title) +
theme(
legend.position = “right”,
panel.background = element_blank(),
axis.line = element_line(colour = “black”, size = 1.1),
legend.background = element_blank(),
legend.text = element_text(size = 12),
legend.title = element_text(size = 14),
legend.key = element_blank(),
axis.text.x = element_text(size = 10),
axis.text.y = element_text(size = 10),
axis.title.x = element_text(size = 14),
axis.title.y = element_text(size = 14)) +
guides(fill = guide_legend(override.aes = list(size = 4))) +
scale_fill_manual(name = group.name, values = color)
le <- ggpubr::get_legend(p)
}else{
x <- pair.pcs[1,i]
y <- pair.pcs[2,i]
p <- ggplot(mapping = aes(
x = pcs[,x],
y = pcs[,y],
fill = group)) +
xlab(paste0(‘PC’, x, ‘ (‘,pc.var[x], ‘%)’)) +
ylab(paste0(‘PC’, y, ‘ (‘,pc.var[y], ‘%)’)) +
geom_point(
aes(fill = group),
pch = 21,
color = strokeColor,
stroke = strokeSize,
size = pointSize,
alpha = alpha) +
scale_x_continuous(breaks = scales::pretty_breaks(n = 5)) +
scale_y_continuous(breaks = scales::pretty_breaks(n = 5)) +
theme(
panel.background = element_blank(),
axis.line = element_line(colour = “black”, size = 1.1),
legend.position = “none”,
axis.text.x = element_text(size = 10),
axis.text.y = element_text(size = 10),
axis.title.x = element_text(size = 14),
axis.title.y = element_text(size = 14)) +
scale_fill_manual(values = color, name = group.name)
}
p <- p + theme(legend.position = “none”)
xdens <- cowplot::axis_canvas(p, axis = “x”)+
geom_density(
mapping = aes(
x = pcs[,x],
fill = group),
alpha = 0.7,
size = 0.2
) +
theme(legend.position = “none”) +
scale_fill_manual(values = color)
ydens <- cowplot::axis_canvas(
p,
axis = “y”,
coord_flip = TRUE) +
geom_density(
mapping = aes(
x = pcs[,y],
fill = group),
alpha = 0.7,
size = 0.2) +
theme(legend.position = “none”) +
scale_fill_manual(name = group.name, values = color) +
coord_flip()
p1 <- insert_xaxis_grob(
p,
xdens,
grid::unit(.2, “null”),
position = “top”
)
p2 <- insert_yaxis_grob(
p1,
ydens,
grid::unit(.2, “null”),
position = “right”
)
pList[[i]] <- ggdraw(p2)
}
pList[[i+1]] <- le
return(pList)
}
小伙伴可以直接运行这两个函数,或者也可以想小云一样整理在一个R文件里面,方便下次使用,
然后我们就可以使用
source(“pca.r”)#载入使用
下面我们就是来看下载入数据的格式,
这是小云自己的数据,小伙伴可以按照形式自行去设置,
df<-read.csv(“pca1.csv” )
df
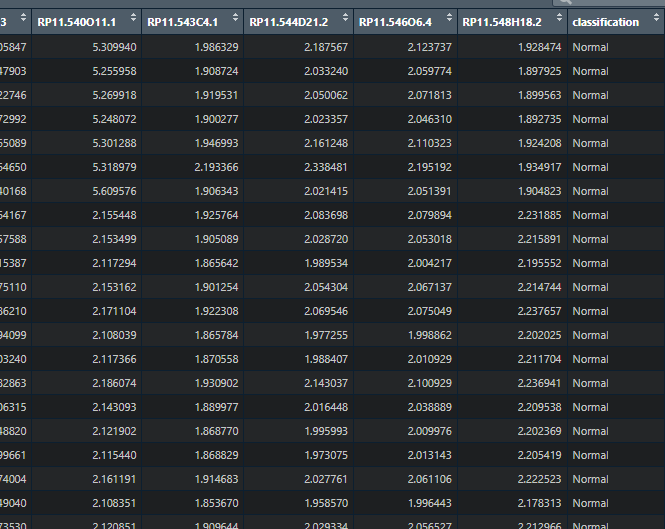
我们展示最重要的一部分,前面都是基因的表达量,纵的表示样本,最后一个我们设置一下分组,作为我们PCA分析的数据形式,就是Classification,小云这里分了四组,文献中分了5组,这个看小伙伴自己的需求,
然后我们计算一下
pca.ncg<-.pca(data = df[,1:8767], ##
is.log = FALSE)
上面这个数字代表基因数量,小云这里就选择这一些
运行完就
 出现,我们就将计算好的值进行可视化,
出现,我们就将计算好的值进行可视化,
.scatter.density.pc(pcs = pca.ncg$sing.val$v[, 1:3], ##展示几个
pc.var = pca.ncg$variation,
strokeColor = ‘gray30’,
strokeSize = .2,
pointSize = 2,
alpha = .6,
title = “A”, ##标题名
group.name = “Stage”, # legned name
group=df$classification, # 选择分组
color=c(“darkorchid4″,”cornflowerblue”,”sandybrown”,”mediumaquamarine”)) -> p#颜色
下面我们使用代码进行展示,
do.call(
gridExtra::grid.arrange,
c(p,ncol=4,nrow=1))
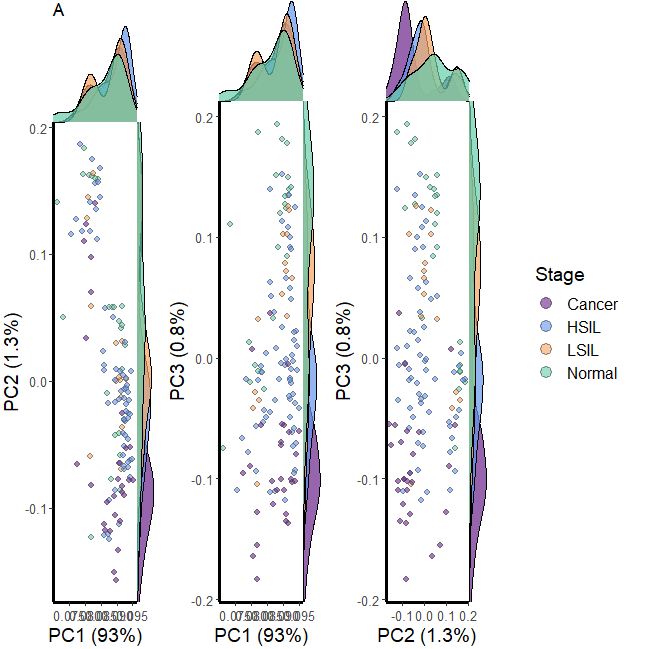
这样一看,和文献中有点不一样,太长了,我们把出图的排列改一下
do.call(
gridExtra::grid.arrange,
c(p,ncol=2,nrow=2))
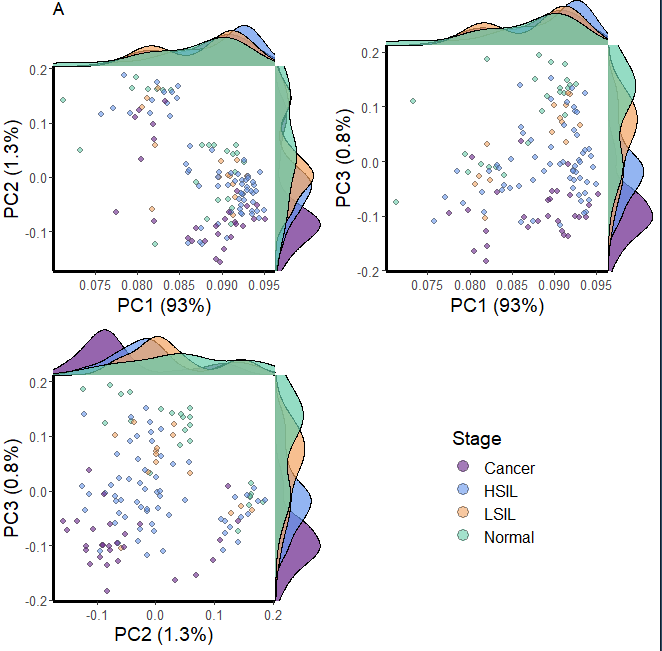
这样一来就差不多了,最后再把点的大小调一下,然后出图,
.scatter.density.pc(pcs = pca.ncg$sing.val$v[, 1:3], ##
pc.var = pca.ncg$variation,
strokeColor = ‘gray30’,
strokeSize = .2,
pointSize = 4,
alpha = .6,
title = “A”, ##
group.name = “Stage”, # legned name
group=df$classification, #
color=c(“darkorchid4″,”cornflowerblue”,”sandybrown”,”mediumaquamarine”)) -> p
do.call(
gridExtra::grid.arrange,
c(p,ncol=2,nrow=2))
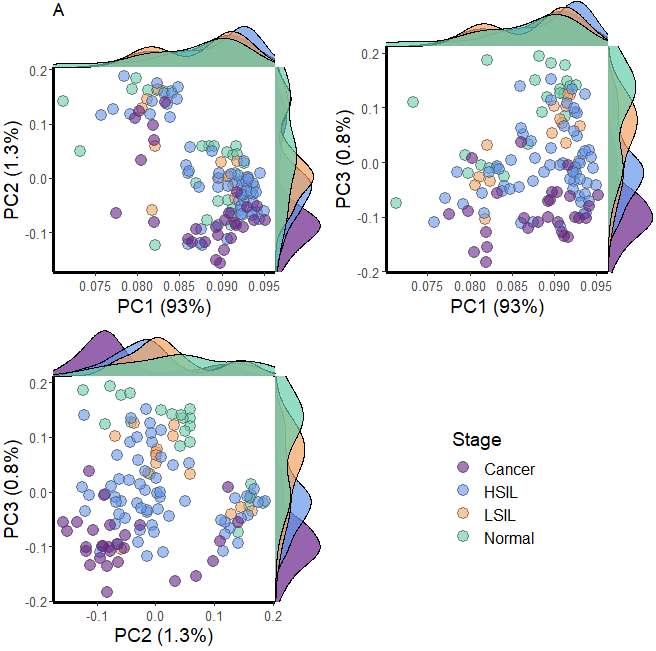
这就一样了基本上就一样了,颜色小云就随机设置的,小伙伴看了有没有心动了,要多多理解代码的意思,上述函数的文件呢,小伙伴需要的可以找我们公众号领取哦
最后呢,小云给小伙伴带个小福利,如果小伙伴觉得自己运行代码太麻烦,可以试试我们的云生信小工具哟,里面有成熟的代码,只要输入合适的数据就可以直接出想要的图呢。
多多关注我们公众号,云生信!
