不用花钱就能学到的GWAS数据通路分析-MAGMA 又赚到了!
点击蓝字
关注小图
随着全基因组关联分析使用样本数量的增加,人们也逐渐认识到很多感兴趣的性状在本质上是由微效多基因控制。通过以有生物学意义的方式整合复杂疾病的信息的基因以及通路分析,是单变异GWAS的有效补充。小图今天将介绍MAGMA,一个基于多变量回归模型的基因以及通路分析工具。
MAGMA的基本分析包括两到三个步骤
-
首先,将snp映射到基因上的注释步骤;
-
第二步,进行基因分析,计算基因p值;
-
第三步,基因水平分析步骤:要么进行广义基因集分析,要么进行基因特性分析,要么两者兼而有之。基因水平分析都是以基因分析为基础的,任何可能的基因分析模型都可以作为基因水平分析的输入。
首先下载MAGMA软件
网址链接:
(https://ctg.cncr.nl/software/magma)
程序本身的下载链接以及一些辅助文件可以在下面找到。对于Linux,二进制文件是在Debian上编译的,但通常也会在其他发行版上运行。如果由于Linux发行版的某些差异而无法找到某些库(通常是关于丢失.dll文件的错误消息),则会提供额外的静态链接二进制文件;使用这个可以解决这个问题。
下面还可以下载MAGMA源代码,如果所提供的二进制文件不支持的话,它可以用来在目标系统上编译程序(注意,适用标准版权;MAGMA的二进制文件和源代码不能发布或修改)。
根据自己需求下载相应版本
注释
注释可通过以下命令执行:
magma --annotate --snp-loc [SNPLOC_FILE] --gene-loc [GENELOC_FILE] --out [ANNOT_PREFIX][SNPLOC_FILE]需要由我们自己提供
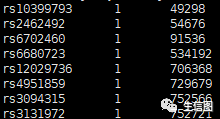
文件包含三列,即前三列为SNP ID, chromosome, 和base pair position
[GENELOC_FILE]可以从MAGMA网站下载,用于不同的构建基因注释参考文件

对于基因定位文件,蛋白质编码基因的基因定位从NCBI位点获得。定位定义为从转录起始位点到转录终止位点的区域。这些位置文件包含Entrez基因id。
基因分析
SNP p值对SNP p值进行基因分析:
magma --bfile [REFDATA] --pval [PVAL_FILE] N=[N] --gene-annot [ANNOT_PREFIX].genes.annot --out [GENE_PREFIX][REFDATA] bfile为原始数据或是参考LD面板,如果数据量不大可以直接使用自己的plink的bed格式原始数据,在原始数据无法获得的时候可以使用magma提供的1000 genome参考数据,biobank级别的数据的情况下,可以随机抽取某个族裔无亲缘关系的一定人数(例如20000人)来构建自己的参考面板。
参考数据文件是从第3阶段的1000个基因组中创建的。数据中的SNP位置参考了人类基因组Build 37。还有一个包含子种群定义的附加文件,可用于从1,000个基因组数据文件中提取特定的子种群。特定于数据的SNP同义词文件包含在数据中。
[PVAL_FILE]由我们自己提供
包含先前计算的SNP P值的文件(在文件[PVAL_FILE]中的“SNP”和“P”列中)。
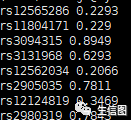
pval为SNP的p值文件,包含两列 SNP 以及 P
[ANNOT_PREFIX]为上一步注释后得到的文件
N为样本量
基因集分析
进行基因集分析:
magma --gene-results [GENE_PREFIX].genes.raw --set-annot [SET_FILE] --out [GS_PREFIX]读取先前执行的基因分析的结果,以及将基因映射到文件[SET_FILE]中指定的基因集(每行对应于一个基因集:基因集的名称后跟基因id,以空格分隔)。
[SET_FILE]为基因集或通路的定义文件,可以直接使用MSigDB下载的文件(注意基因ID要与之前相对应)
默认输出三个.gsa.out .gsa.genes.out, .gsa.sets.genes.out
.gsa.out 为最主要的输出文件,包含了各个通路的检验结果
gsa.out 包含了各个通路的检验结果
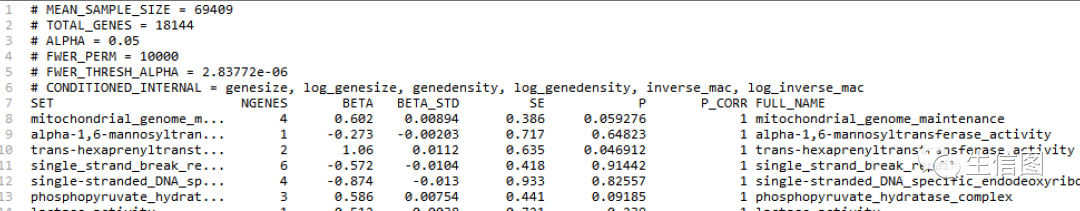
gsa.genes.out 基因关联结果
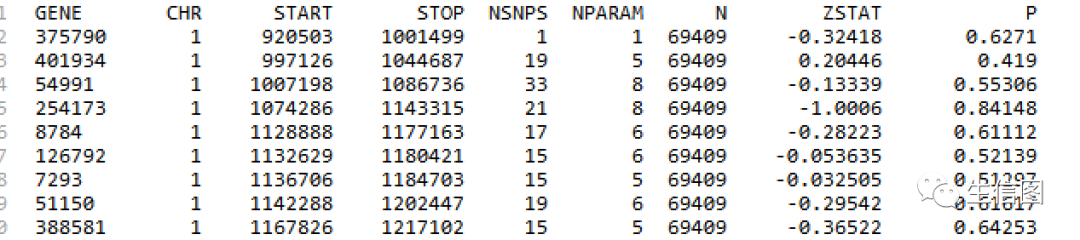
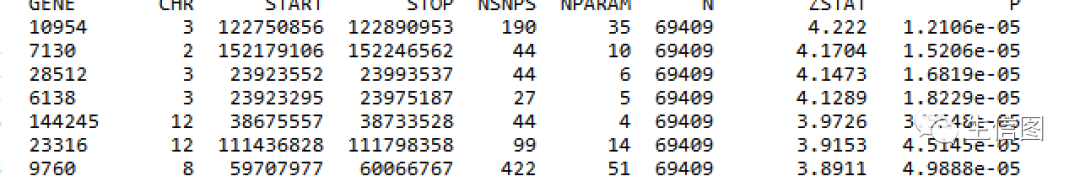
gsa.sets.genes.out 多重检验调整后, 显著的通路里的基因的信息

有疑问欢迎咨询小图的微信公众号(生信图)和云生信生物信息学平台( http://www.biocloudservice.com/home.html)
欢迎使用:云生信平台 ( http://www.biocloudservice.com/home.html)

|
往期推荐 |
|
|
|
|
|
|
👇点击阅读原文进入网址