惊掉下巴!你不知道的ATAC-seq重复样本处理方式
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

小果课堂随机提问:小伙伴们知道重复样本应该怎么样处理吗?
下面是一些基础知识:
重复样本的处理方式是指在数据分析中,如何处理同一组或同一样本的多次测量或分析结果。重复样本的处理方式可能因为不同的研究目的和数据特征而有所不同,但一般可以分为以下几种:
1. 样本重复性检验:通过计算样本间的相关系数、绘制样本聚类图或主成分分析图等方法,评估组内样本的重复性是否良好,是否有离群样本需要剔除。
2. 样本过抽样或欠抽样:当数据存在不均衡问题时,可以通过增加少数类样本或减少多数类样本的方法,使数据更加均衡。
3. 样本权重调整:当数据存在不均衡问题时,也可以通过给少数类样本赋予更高的权重或给多数类样本赋予更低的权重的方法,使数据更加均衡。
4. 样本混合或平均:当数据存在较大的个体差异或背景波动时,可以通过将多个样本混合作为一个生物学重复或对多个技术重复取平均值的方法,降低数据的噪声。
上面是一些常见的重复样本的处理方式,具体应用时需要根据实验设计和数据特点进行选择和优化。
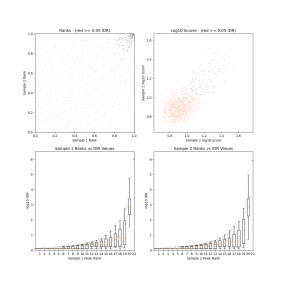
小果最近沉迷ATAC-seq,那就以ATAC-seq当例子来教大家重复样本的处理方式吧。针对ATAC-seq数据,要求必须有2次或更多次生物学重复(十分珍贵或者稀有样本除外,但必须做至少2次技术重复)。理论上重复样本的peaks应该有高度的一致性,实际情况并不完全与预期一致。如何评价重复样本的重复性的好坏?如何得到一致性的peaks?
上面的几个问题是不是又给小伙伴们整晕了呢?下面小果教大家使用IDR来处理ATAC-seq中的重复样本
首先是IDR软件的下载
IDR的安装十分简单,它的安装方法有多种,最简单的一种是使用 conda 命令:
conda install -c bioconda idr这样可以自动安装 idr及其所有的 python 依赖项
另一种方法是使用 pip 命令:
pip install idr这样也可以自动安装 idr及其所有的 python 依赖项。不过这里小果要建议小伙伴们使用IDR时,在MACS2 callpeak的时候参数不要设置太严格哦,这样才能鉴定出更多的peak,并且使用IDR需要先对MACS2的结果文件narrowPeak根据-log10(p-value)进行排序。
下面是小果的代码:
# 参数设置可以适当放宽macs2 callpeak -t test.final.bam -n sample --shift -100 --extsize 200 --nomodel -B --SPMR -g hs 2> sample.macs2.log# 对peak 按照 -log10(p-value) 排序sort -k8,8nr sample_peaks.narrowPeak > sample_peaks.sort.narrowPeak
下面可以直接使用idr 来处理重复样本。
小果的代码是这样的:
idr --samples Rep1_sorted_peaks.narrowPeak Rep2_sorted_peaks.narrowPeak--input-file-type narrowPeak--rank p.value--output-file idr--plot--log-output-file sample.idr.log
下面是一些参数的解释:
1. –samples: 输入文件
2. –input-file-type:输入文件格式
3. –rank p.value:以p-value排序
4. –output-file: 输出文件路径
5. –plot:绘制结果图
小果提醒大家,idr每次只能一次处理两个样本哦
这是小果的结果图,是不是很直观呢
今天的ATAC-seq中重复样本处理的学习就到这里啦,感兴趣的小伙伴可以找小果讨论哦,我们明天见咯~

点击“阅读原文”立刻拥有
↓↓↓