今日神器devtools:一键安装github上所有生信工具包
收录于话题
功能介绍
生信入门、R语言、生信图解读与绘制、软件操作、代码复现、生信硬核知识技能分享、服务器等….
Hi!小果今天来教大家如何使用一个神奇的R包:devtools。下面小果将为大家介绍devtools包的基本功能和用途,以及如何实战应用,感兴趣的同学就和小果一起学习吧!

devtools是何方神圣
devtools是R语言中很有用的一个包,它可以直接从GitHub上下载和安装其他R包及其依赖关系。在生物信息学研究中,我们经常需要使用很多不同的生物信息学软件包来进行数据分析,例如进行基因组学和转录组学分析时需要使用DESeq2、edgeR等分析包;进行进化树分析需要使用ape、phangorn等包;进行函数预测和基因组定位需要使用hmmr和blast等包。而这些包可能还依赖其他一些基础包,如果直接在R中安装可能会因为依赖关系问题等各种原因而安装不成功。此时使用devtools就可以很方便地一次性从GitHub直接下载并安装这些包及其所有依赖关系。除此之外”devtools” 也是一个在R语言中广泛使用工具,它提供了丰富的功能和函数,用于帮助你创建、测试和发布你的R包。
以下是”devtools”的一些主要功能:
创建R包:devtools提供的create()函数可用于创建新的R包,包括自动创建文件夹结构和必要的文件。
测试R包:devtools提供的test()函数能够运行你的R包中的所有测试,并提供有用的反馈。
发布R包:devtools能够帮助你将你的R包发布到CRAN或其他平台,例如:Github。
加载和重新加载R包:devtools可以帮助你在开发过程中快速加载或重新加载你的R包。
安装及常用功能介绍
1
安装及加载devtools包
install.packages(“devtools”) # 安装方法
library(devtools) # 加载包
2
创建一个新的R包
devtools::create(“my_new_package”) # 创建一个名为my_new_package的新包
3
构建R包
build(“mypackage”) # 构建一个名为my_new_package的新包
4
测试R包
devtools::test(“my_new_package”) # 测试my_new_package包
5
发布R包
devtools::release(“my_new_package”) # 发布my_new_package包
6
安装R包
install_github(“xiaoguo/my_new_package”) # 使用install_github()函数安装R包
7
加载和重新加载R包
devtools::load_all(“my_new_package”) # 加载或重新加载my_new_package包
小果真心话
实不相瞒,小果使用devtools最多的情况就是下载R包,有些R包只能从github上下载,有如下几个原因:
1.R包的开发者没有将其提交到CRAN或其他官方的源,而是选择在github上进行开发和维护。这样可以方便地使用git进行版本控制和协作,也可以让用户更快地获取最新的更新和修复。
2.R包的开发者已经将其提交到CRAN或其他官方的源,但由于某些原因,该源没有及时更新或同步该R包的最新版本。这样就导致用户在该源上无法找到或安装该R包,而只能从github上下载。
3.R包的开发者已经停止了对该R包的开发和维护,而只在github上保留了该R包的历史版本。这样就使得用户无法在其他源上找到或安装该R包,而只能从github上下载。
这让许多生信小白抓耳挠腮,无法通过常规方法下载。
示例教学
下面小果就以DESeq2包的下载为例,带大家感受devtools下载R 包的快乐。我们先使用install.packages(‘DESeq2’)函数下载,会出现报错。

下面使用devtools从github安装:
install_github(repo,ref = "HEAD",subdir = NULL,auth_token = github_pat(quiet),host = "api.github.com",dependencies = NA,upgrade = c("default", "ask", "always", "never"),force = FALSE,quiet = FALSE,build = TRUE,build_opts = c("--no-resave-data", "--no-manual", "--no-build-vignettes"),build_manual = FALSE,build_vignettes = FALSE,repos = getOption("repos"),type = getOption("pkgType"),...)
参数解释
repo: 要安装的Github仓库的名称,必须为字符向量类型。
ref: 指定从Github中安装哪个分支或标签,默认为最新版本。
subdir: 仓库中的子目录名称(可选)。
host: 使用Github镜像,默认为github.com。
dependencies: 指定要安装的依赖关系,可选项包括NA(默认,自动安装所有必需的依赖项)、”Depends”(仅安装必需的依赖项)和”Imports”(安装必需和导入的依赖项)。
upgrade: 指定是否更新已安装的软件包。可选项包括:”default”(默认,只在必要时更新)、”ask”(询问用户是否要更新)、”always”(总是更新)和”never”(永不更新)。也可以使用字符串”bioc”,在安装bioconductor包时,会强制更新。
force_deps: 指定是否强制重新安装依赖项。默认为FALSE。
build_vignettes: 是否在安装时构建vignettes。如果不指定,则根据全局选项构建。
build_opts: 一个字符向量,指定在构建软件包时要传递给R CMD build的选项。
build: 是否在安装时构建源代码包。默认为TRUE。
build_manual: 是否构建PDF手册(如果存在)。
quiet: 是否静默安装。
build_args: 传递给R CMD build和R CMD install的其他构建参数。
repos: 要使用的仓库类型和地址。
type: 安装的软件包类型,通常自动检测。
…: 其他参数,包括安装时的额外命令行选项。
从github安装DESeq2包的代码示例:
install.packages("devtools")library(devtools)install_github("mikelove/DESeq2")
#devtools会自动从
https://github.com/mikelove/DESeq2这个仓库(如图)下载最新的DESeq2包及其所有需要的依赖包,一键完成安装
安装成功
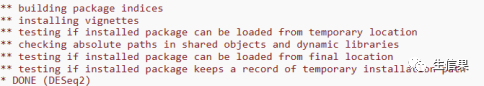
第一次载入会加载许多依赖包,要消耗一点时间。
library(DESeq2)
总结
总之,在进行生物信息学研究时,使用devtools直接从GitHub一键安装依赖完整的R包及其最新版本,可以大大简化安装步骤,提高工作效率。它堪称生物信息学家必不可少的一个利器。
以上就是对devtools包的基本介绍和使用方法。掌握了这些方法,你就可以更方便地创建、测试、发布、下载和管理你的R包了。你可以根据自己的需求和情况,对这些代码进行修改和扩展!
怎么样,你学会了吗?小果今天的分享就到这里,欢迎大家和小果一起讨论学习哦!我们下期再见~
往期推荐

