靶基因怎么预测转录因子关系?快来试试这个!
点击蓝字 关注我们
小伙伴们大家好啊,大海哥又和大家见面了,今天大海哥要介绍一个数据库以及一个插件,首先介绍一下JASPAR数据库(https://jaspar.genereg.net/),这个数据库大家应该有所耳闻,它包含了9个不同的子库,其中CORE库属于高质量,非冗余转录因子数据库,包含的信息源于已经实验证实的真核生物转录因子结合位点。可供查找的物种有脊椎动物,线虫,昆虫,真菌和植物。
今天我们主要讲解如何使用这个数据库。首先看看主页是什么样子的

那么展开操作之前,我们先了解一下,转录因子是什么?
转录因子(Transcription Factor)其实是一类蛋白质,它们在基因表达调控中起着重要的作用。转录因子能够结合到DNA的特定序列上,并调控基因的转录过程,从而控制蛋白质的合成。转录因子通过与DNA上的特定序列结合,调控基因的表达。这些特定序列称为转录因子结合位点(Transcription Factor Binding Sites),通常位于基因的启动子区域。转录因子的结合可以促进或抑制基因的转录,从而影响蛋白质的产生。
介绍完,开始操作吧,第一次使用的小伙伴还可以点击下方图示的位置JASPAR interactive tour,会有流程指导哦!
首先点击左侧工具栏中search,可以查找感兴趣的数据。可以通过TF名称或ID、物种、分类单元、uniprot ID或任何其他关键字进行搜索。Advanced Options为高级查询,以检索人的转录因子SPI1为例,下方可选框中选择筛选要求,快速锁定目标数据。

点击Search后,查询到了6条结果

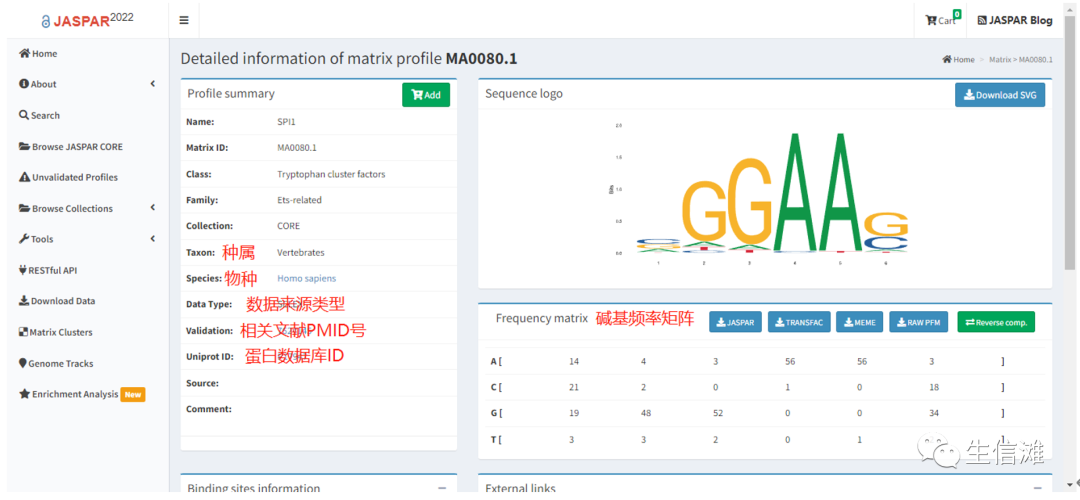
点击ID号,可以跳转至SPI1 motif详情,包括文件摘要、序列logo图、PFM矩阵(文件可下载)、TF-binding信息(FASTA格式为该motif在基因组范围的结合区域的序列,bed格式为该motif在基因组范围的结合位点,可点击下载)、外部链接、版本信息、ChIP-seq centrality、TFFM和其它信息
除此之外呢,我们还可以使用Cytoscape中的iregulon插件来预测靶基因关联的转录因子哦!
首先我们需要打开Cytoscape,点击上方的Apps

然后搜索iRegulon,稍等一会就会跳转到下载页面

点击我们需要的软件后,注意一下我们使用的版本是不是符合要求的,如果不符合的话,是没有办法安装的,大家要注意!
点击安装后,稍等一会回到Cytoscape界面,我们就可以看到Installed apps这里出现了iRegulon

然后我们回到Cytoscape主页面,上传我们需要分析的靶基因
只用一列就可以了

就像这样就可以,然后点OK
再选中所有的基因

就像这样全部显示黄色就可以,然后点击上面的Apps,按下面的步骤操作就可以开始分析啦
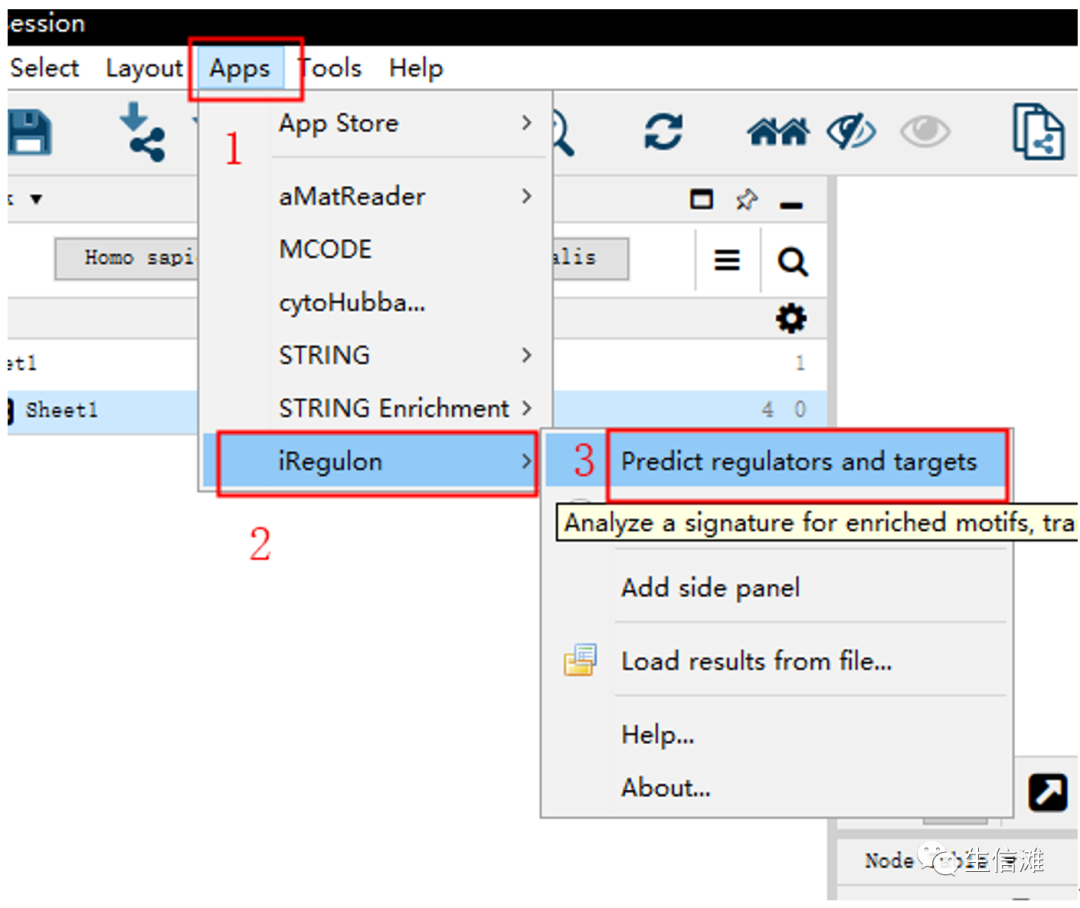
就会出现如下所示

这里有几个关键指标
Node information
Number of selected genes:所预测的基因数目
Ranking
Type of search space:基于基因
Motif collection: 10k/6k
Track collection:1120 ChlP-se/750
Putative regulatory region:起始位点上下游端
Motif/Track rankings database:排序所根据的数据库
Recovery:
富集分数
AUC值
排序阈值
TF prediction
FDR:发现错误率
最后结果就是包含下图三部分Motifs、Tracks和Transcription Factors(TF)

如果对某个TF感兴趣,想画个网络图看看,可以点击Transcription Factors,然后选中TF,点左上角的加号按钮就可以了

然后就会出现下图所示的网络图哦!

怎么样,是不是很方便,不仅如此,还可以通过TF预测对应靶基因呢!点击下图位置

然后会出现如下的界面

然后可以选择物种和数据库,点击Submit,就可以出现网络图啦!

是不是很好用!小伙伴们如果方便,不妨自己动手试一试哦!说不定会有很好的结果图哦!(最后推荐以下大海哥新开发的零代码云生信分析工具平台,包含超多零代码小工具,上传数据一件出图,感兴趣的小伙伴欢迎来参观哟,网址:http://www.biocloudservice.com/home.html)
点击“阅读原文”进入网址