10×genomics单细胞测序分析(一)环境搭建与Cell Ranger数据处理
点击蓝字 关注我们
10×genomics因其分选细胞效果好,通量高等优点已成为主流的单细胞测序平台。
大海哥上期对它的原理进行了介绍,感兴趣的小伙伴可以去看。这期大海哥将介绍10×genomics单细胞测序分析环境的搭建与用10×平台官方出的数据处理流程Cell Ranger进行数据处理与count表达矩阵的生成。
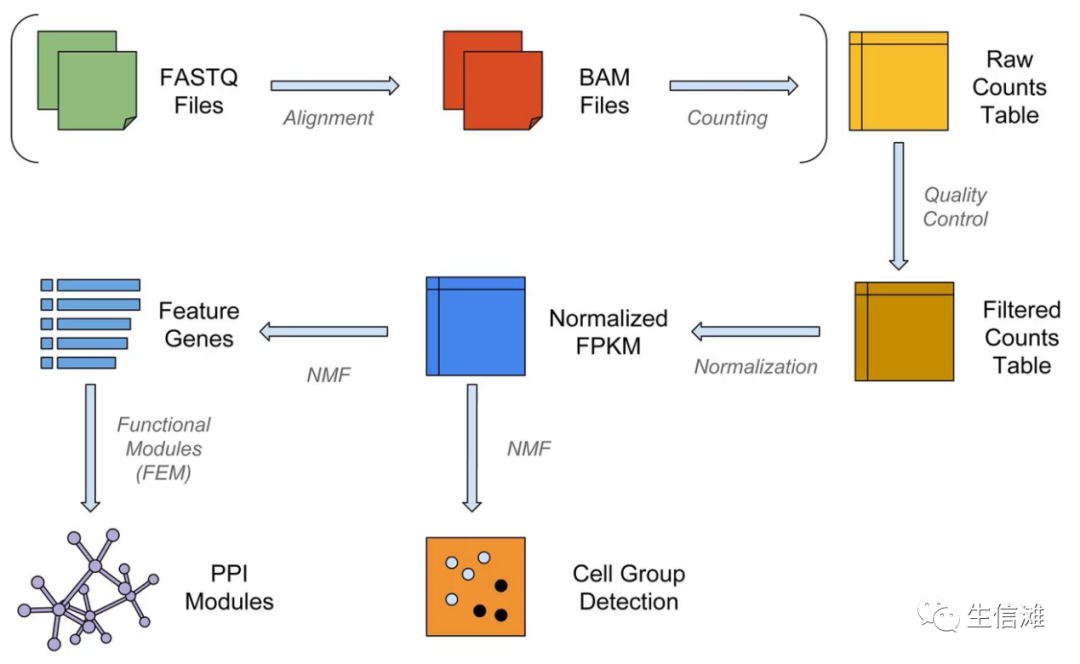
通常这是单细胞分析的整个流程。
与许多组学分析一样,先将FASTQ文件比对,转换为二进制的BAM文件,然后进行基因表达计数。拿到表达矩阵后才能进行下一步的分析。
今天大海哥带大家完成流程的搭建与上游的的数据处理。就是这张图上面三个步骤。
一、准备工作
(一)软件安装
1.CellRanger
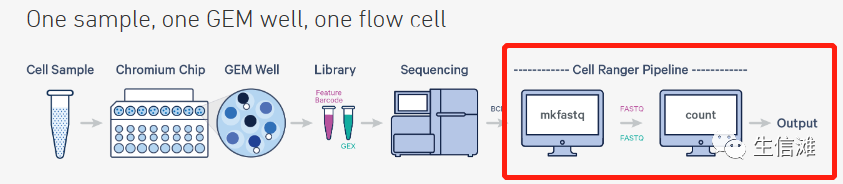
Cell Ranger是10×genomics官方出的数据处理分析工具,它的适配度好,功能强大,操作简单等优点,成为了单细胞测序数据分析的必备工具。
在获得单细胞的测序数据后,使用Cell Ranger来进行数据的处理与分析的工作流程。
What is Cell Ranger? -Software -Single Cell Gene Expression -Official 10x Genomics Support
上面是10×genomics测序平台Cell Ranger主页链接,详细介绍了Cell Ranger是什么与怎么用。
下载需要填写信息完成注册。
复制下载链接到linux命令行,运行并下载

#随后解压缩下载的软件包
tar -zxvf cellranger-7.1.0.tar.gz
在命令行敲一下,ok,软件安装好了。
2.Loupe Browser
Loupe Browser是一个桌面应用程序,提供交互式可视化分析来自不同 10x 基因组学解决方案的数据的功能。它能轻松查询 10x 基因组学数据的不同视图快速深入了解数据。
下载仍然需要填写一次信息。填写好信息后便可以下载。
在windows系统下打开安装包,进行安装即可。
这是应用的界面
3.bcl2fastq
是illumina平台内置的一个软件,Cell Ranger没有集成这个软件,我们需要自己去下载。有了这个软件之后,Cell Ranger可以完成从bcl测序文件,到最后结果生成,一条龙式地完成。
bcl2fastqandbcl2fastq2Conversion Software Downloads (illumina.com.cn)
bcl2fastq的网站
同样需要注册填写信息,就可以进一步安装了
https://support.illumina.com.cn/content/dam/illumina-support/documents/downloads/software/bcl2fastq/bcl2fastq2-v2-18-0-12-tar.zip这里贴上下载的地址
unzip bcl2fastq/bcl2fastq2-v2-18-0-12-tar.ziptar -zxvf bcl2fastq/bcl2fastq2-v2-18-0-12-tar.gz
#编译软件
cd bcl2fastq2chmod ugo+x src/configurechmod ugo+x src/cmake/bootstrap/installCmake.shmkdir bincd bin../src/configure --prefix=${PWD}/binmakemake install
4.创建conda环境安装软件
#安装mambaconda install -y mamba#创建虚拟环境conda crate -n -y singlecell#激活环境conda activate singlecell#下载软件mamba install -y fastqcmamba install -u multiqcmamba install -y fastpmamba install -y starmamba install -y bioconductor-singlermamba install -y samtoolsmamba install -y featurecountsmamba install -y r-seuratmamba install -y umi_tools
二、数据下载
1.参考序列下载
#人基因组wget https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-GRCh38-2020-A.tar.gz#小鼠基因组wget https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-mm10-2020-A.tar.gz#人和小鼠混合wget https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-GRCh38-and-mm10-2020-A.tar.gz#Sample Index Set Sequenceswget https://cf.10xgenomics.com/supp/cell-exp/chromium-shared-sample-indexes-plate.csvwget https://cf.10xgenomics.com/supp/cell-exp/chromium-single-cell-sample-indexes-plate-v1.csvwget https://cf.10xgenomics.com/supp/cell-exp/gemcode-single-cell-sample-indexes-plate.csv
2.练习数据下载
#pbmc 1k数据wget https://cf.10xgenomics.com/samples/cell-exp/3.0.0/pbmc_1k_v3/pbmc_1k_v3_fastqs.tar1000个单细胞的测序数据
三、使用cellranger分析单细胞数据
(一)Basecalling
1.将测序原始数据bcl文件转换为fastq文件
#案例数据,illuminca测序原始数据wget https://cf.10xgenomics.com/supp/cell-exp/cellranger-tiny-bcl-1.2.0.tar.gzwget https://cf.10xgenomics.com/supp/cell-exp/cellranger-tiny-bcl-simple-1.2.0.csvtar -zxvf cellranger-tiny-bcl-1.2.0.tar.gz

这是这个原始测序数据所包含的内容,数据在这个Data目录下面。
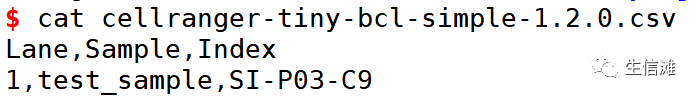
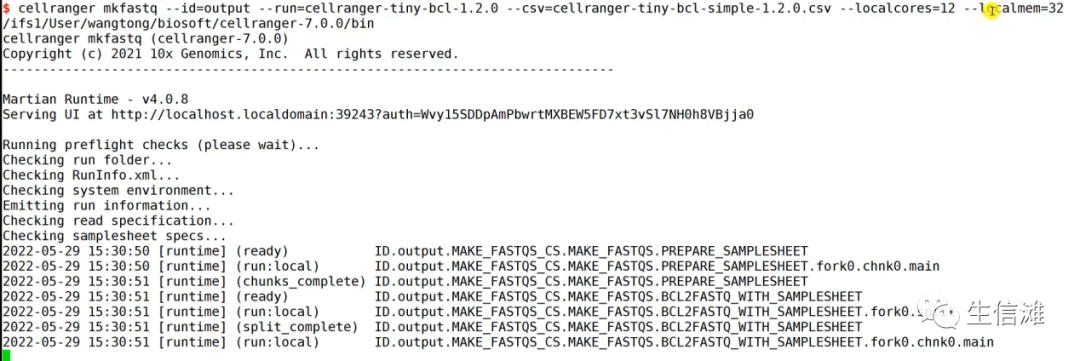
非常简单,只要输入文件,及输出的文件夹就行了。因为数据很小,很快就运行好了。
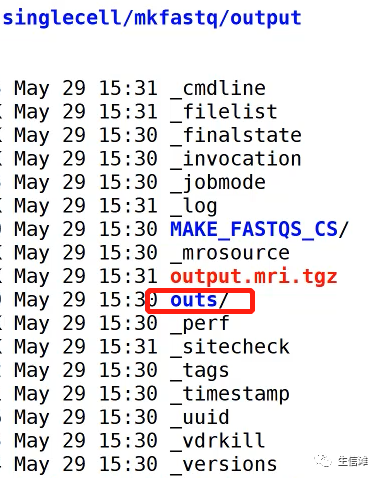
生成的fastq就在这个文件夹里。
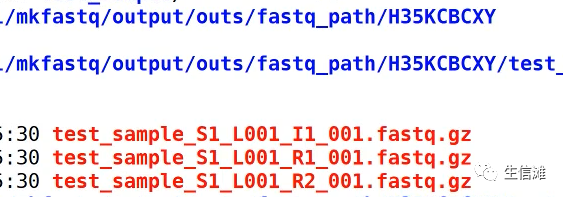
Test_sample就是最终的结果。文件名会根据simple文件的名称命名。
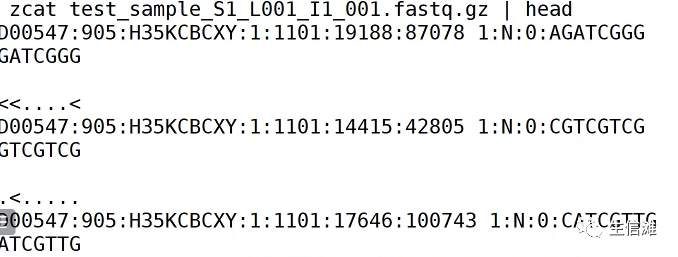
打开第一个看一下,其为Index序列。
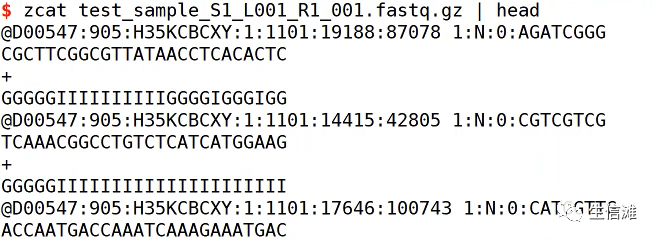
Read1为barcod+UMI
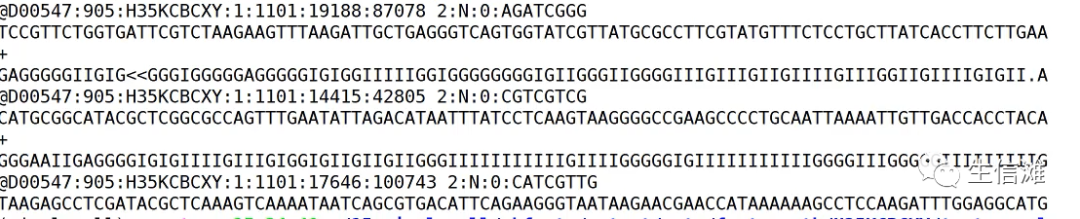
Read2是真正的我们想要的测序序列。
该部分为演示如何将bcl转化为fastq。
2.质控
接下来我们对练习数据进行处理,这里我们打开案例的数据。使用通配符*匹配所有fastq的read2。然后进行质控。
#对read2 进行质控ll pbmc_1k_v3_fastqs/pbmc_1k_v3*_R2_001.fastq.gzfastqc -t 12 -f fastq -o qc1 pbmc_1k_v3_fastqs/pbmc_1k_v3*_R2_001.fastq.gz
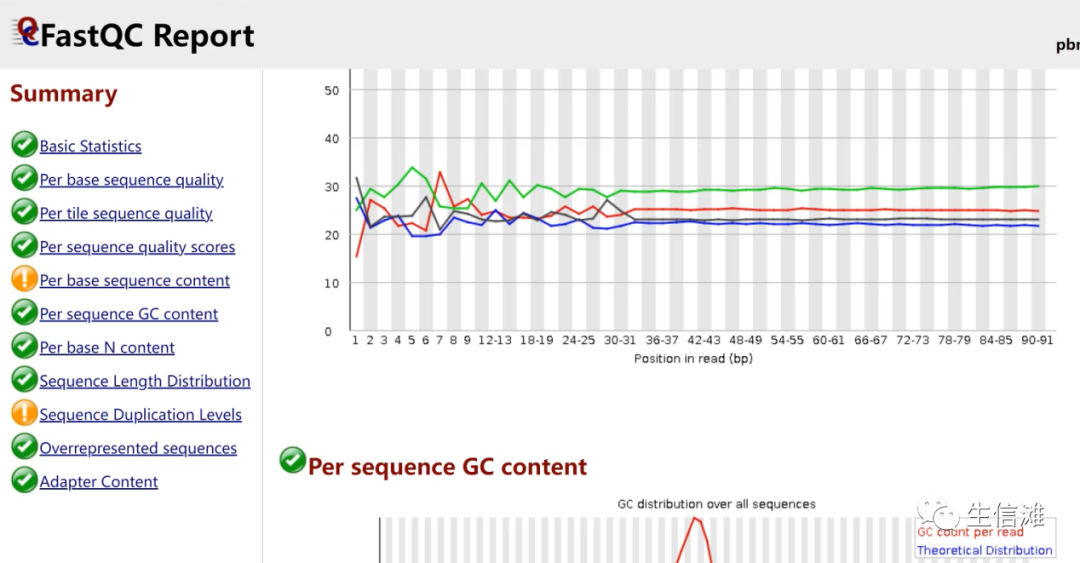
质控结果,显示测序数据质量较好。
10×genomics的测序数据通常质量较好,所以绝大部分不用进行质控以及过滤。
实在过滤的话呢,由于read1和read2不一样,前者是barcode,后者是测序数据,对测序数据过滤掉以后,二者索引不匹配,引起操作麻烦。
(二)生成表达矩阵
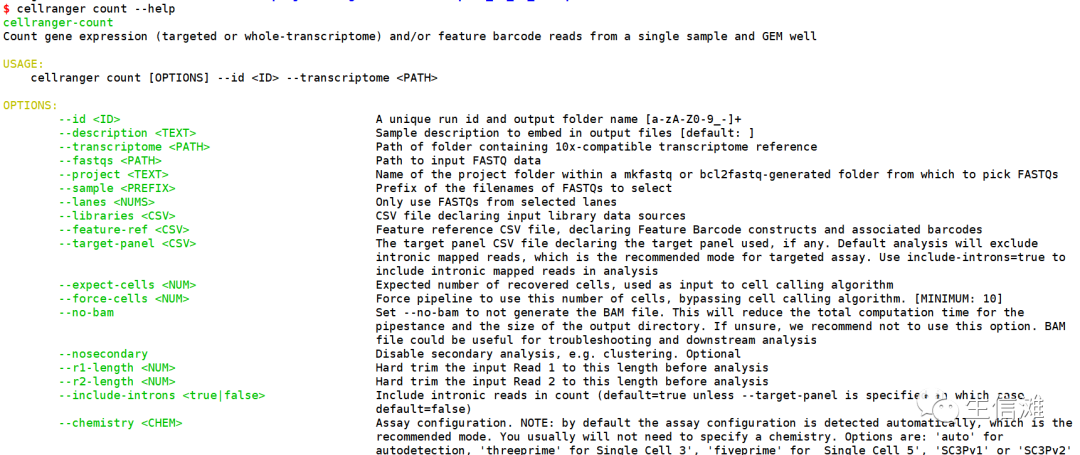
Cellranger count的选项参数,该软件主打的就是一个简单方便,用起来简单绝对是个优点。
输入前面三个参数就可以工作了。
#计算count 矩阵,pbmc 1000个细胞,等号之间不要有空格cellranger count --id=run_count_1kpbmcs --fastqs=pbmc_1k_v3_fastqs --sample=pbmc_1k_v3--transcriptome=refdata-gex-GRCh38-2020-A/--localcores=12 --localmem=32
这样它就运行起来了。
最终的结果在outs里面
大海哥今天的讲解就到这里了~下期我将会带来对单细胞测序分析结果的解读~我们不见不散

生信滩公众号

点击“阅读原文”进入网址
