三分钟绘制双曲线新型火山图
收录于话题
生信人R语言学习必备
立刻拥有一个Rstudio账号
开启升级模式吧
(56线程,256G内存,个人存储1T)

双曲线火山图
最近在很多的文章中出现了下图类似的新型火山图,其实原理就是,分别以原来的的阈值线为基准,画两个原始函数为y=1/x的的曲线。这样对基因进行分类,有利于筛选差异更显著的基因。因此图形也变的美观。

安装相应的包并加载
相关R包载入:library(dplyr)library(ggplot2)library(ggrepel)library(patchwork)本地测试数据读入,具体数据见附件df <- read.csv('./test.xls',header = T,sep = "t")head(df)#筛选阈值确定,一般阈值是p<0.05,|log2FC|>1pvalue = 0.05log2FC = 1根据阈值添加上下调分组标签:group <- case_when(log2FC > log2FC & df$Pvalue < pvalue ~ "up",log2FC < -log2FC & df$Pvalue < pvalue ~ "down",TRUE ~ 'none')head(df)转换为因子指定绘图顺序;'-log10(pvalue)' <- -log10(df$Pvalue) #新增-log10p列group <- factor(df$group, levels = c("up","down","none"))ggplot2建立映射:p <- ggplot(data = df,aes(x = log2FC, y = `-log10(pvalue)`, color = group)) + #建立映射geom_point(size = 2.2) #绘制散点p

#根据自己的需求可以调整下列参数,进行绘图。p1 <- p +scale_x_continuous(limits = c(-7, 7), breaks = seq(-7, 7, by = 2))+scale_y_continuous(expand = expansion(add = c(0.5, 0)),limits = c(0, 10), breaks = seq(0, 10, by = 5))

#自定义颜色与主题:mycol <- c("#EB4232","#2DB2EB","#d8d8d8")mytheme <- theme_classic() +theme(axis.title = element_text(size = 15),axis.text = element_text(size = 14),legend.text = element_text(size = 14))p2 <- p1 +scale_colour_manual(name = "", values = alpha(mycol, 0.7)) +mytheme

#添加阈值辅助线p3 <- p2 +geom_hline(yintercept = c(-log10(pvalue)),size = 0.7,color = "black",lty = "dashed") + #水平阈值线geom_vline(xintercept = c(-log2FC, log2FC),size = 0.7,color = "black",lty = "dashed") #垂直阈值线
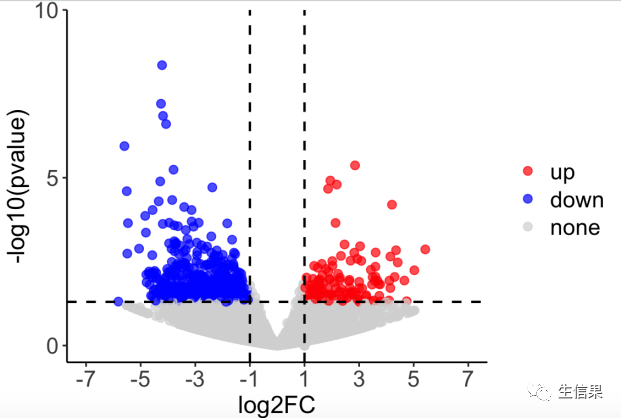
添加双曲线
#根据反比例函数 y = 1/X和自己设定的阈值自定义双曲线函数f <- function(x){inputx <- seq(0.0001, x, by = 0.0001)y <- 1/(inputx) + (-log10(pvalue))dff <- rbind(data.frame(x = inputx + log2FC, y = y),data.frame(x = -(inputx + log2FC), y = y))return(dff)}#根据函数生成所需的曲线数组坐标:dff_curve <- f(2)head(dff_curve)#需要新增曲线数值列:##每列log2FoldChange值在曲线上对应的y轴坐标;df$curve_y <- case_when(df$log2FC > 0 ~ 1/(df$log2FC-log2FC) + (-log10(pvalue)),df$log2FC <= 0 ~ 1/(-df$log2FC-log2FC) + (-log10(pvalue)))#跟前文讲述的一样根据曲线新增上下调分组标签:df$group2 <- case_when(df$`-log10(pvalue)` > df$curve_y & df$log2FC >= log2FC ~ 'up',df$`-log10(pvalue)` > df$curve_y & df$log2FC <= -log2FC ~ 'down',TRUE ~ 'none')#绘制新双曲线阈值火山图:df$group2 <- factor(df$group2, levels = c("up","down","none")) #指定顺序mycol2 <- c("#F8B606","#4A1985","#d8d8d8")p4 <- ggplot(data = df,aes(x = log2FC, y = `-log10(pvalue)`, color = group2)) + #使用新的分组geom_point(size = 2.2) +scale_x_continuous(limits = c(-7, 7), breaks = seq(-7, 7, by = 2)) +scale_y_continuous(expand = expansion(add = c(0.5, 0)),limits = c(0, 10), breaks = seq(0, 10, by = 5)) +scale_colour_manual(name = "", values = alpha(mycol2, 0.7)) +geom_line(data = dff_curve,aes(x = x, y = y), #曲线坐标color = "black",lty = "dashed", size = 0.7) +mythemep4

添加标签
#筛选显著性top10标签:top10 <- filter(df, group2 != "none") %>%distinct(GeneName, .keep_all = T) %>%top_n(10, abs(log2FC))p5 <- p4 +geom_text_repel(data = top10,aes(x = log2FC, y = `-log10(pvalue)`, label = GeneName),force = 80, color = 'black', size = 3.2,point.padding = 0.5, hjust = 0.5,arrow = arrow(length = unit(0.02, "npc"),type = "open", ends = "last"),segment.color="black",segment.size = 0.3,nudge_x = 0,nudge_y = 1)p5

补充关于火山图差异筛选的统计学意义:
1、什么是fold change?
差异倍数,简单来说就是基因在一组样品中的表达值的均值除以其在另一组样品中的表达值的均值。所以火山图只适合展示两组样品之间的比较。
2、为什么要做Log 2转换?
绘制到图中时,上调占的空间多,下调占的空间少,展示起来不方便。所以一般会做Log 2转换。默认我们都会用两倍差异 (fold change == 2 | 0.5)做为一个筛选标准。
3、P-value都比较熟悉,统计检验获得的是否统计差异显著的一个衡量值,约定成俗的P-value<0.05为统计检验显著的常规标准。
4、什么是adjusted P-value?
做差异基因检测时,要对成千上万的基因分别做差异统计检验。统计学家认为做这么多次的检验,本身就会引入假阳性结果,需要做一个多重假设检验校正。
5、为什么做-Log 10转换呢?
因为FDR值是0-1之间,数值越小越是统计显著,也越是我们关注的。-Log 10 (adjusted P-value)转换后正好是反了多来,数值越大越显著,而且以10为底很容易换算回去。
如果遇到不懂的也可以借助线上的云平台哦~http://www.biocloudservice.com/home.html
好了今天的火山图就讲到这里,欢迎大家有问题与小果一起讨论哦~
其他相关分析内容,例如预测肿瘤样本药物敏感性分析(http://www.biocloudservice.com/712/712.php),预测某样本亚型对免疫治疗的反应(http://www.biocloudservice.com/292/292.php),单样本富集算法分析免疫浸润丰度(http://www.biocloudservice.com/106/106.php),计算64种免疫细胞相对含量(http://www.biocloudservice.com/107/107.php)等都可以用本公司新开发的零代码云平台生信分析小工具,一键完成该分析奥,感兴趣的小伙伴欢迎来尝试奥,网址:http://www.biocloudservice.com/home.html。今天小果的分享就到这里,下期在见奥。
小果友情推荐
好用又免费的工具安利



点击“阅读原文”立刻拥有
↓↓↓