难以想象!系统发育树也可以惊艳众人!ggtreeExtra包,让你的数据化身“绚丽多彩”的系统发育树!


公众号后台回复“111”
领取本篇代码、基因集或示例数据等文件
文件编号:240402
需要租赁服务器的小伙伴可以扫码添加小果,此外小果还提供生信分析,思路设计,文献复现等,有需要的小伙伴欢迎来撩~

if (!require("BiocManager", quietly = TRUE))install.packages("BiocManager ")BiocManager::install("ggtreeExtra") # 在BiocManager环境下安装ggtreeExtra查看是否安装成功packageVersion("ggtreeExtra") # 查看ggtreeExtra版本

BiocManager::install("ggtree") # 在BiocManager环境下安装ggtreeBiocManager::install("ggplot2") # 在BiocManager环境下安装ggplot2BiocManager::install("ggnewscale ") # 在BiocManager环境下安装ggnewscaleBiocManager::install("ggstar") # 在BiocManager环境下安装ggstar
library(ggtreeExtra) # 载入ggtreeExtra包library(ggtree) # 载入ggtree包library(ggplot2) # 载入ggplot2包library(ggnewscale) # 载入ggnewscale包library(ggstar) #载入ggstar包
set.seed(1024) # 设置随机参数tr <- rtree(100) # 随机生成一个包含一个节点的树dt <- data.frame(id=tr$tip.label, value=abs(rnorm(100)), group=c(rep("A",50),rep("B",50))) #随机生成一个数据框,包含一百个随机值,分成AB两组df <- dt # 赋值到变量dfdtf <- dt # 赋值到变量dftcolnames(df)[[3]] <- "group2" # 给第三列命名colnames(dtf)[[3]] <- "group3" # 给第三列命名p <- ggtree(tr, layout="fan", open.angle=0) # 绘制最里层的树形结构p # 显示树形结构

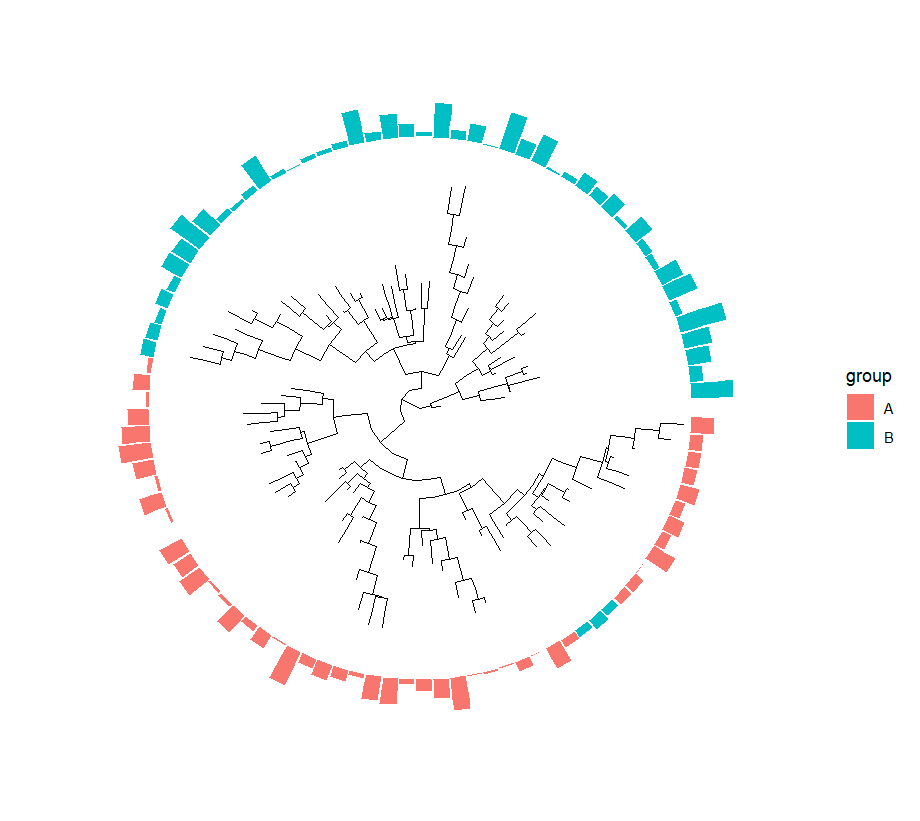
p1 <- p +geom_fruit(data = dt,geom = geom_col,mapping = aes(y = id,x = value,fill = group)) +new_scale_fill()p1
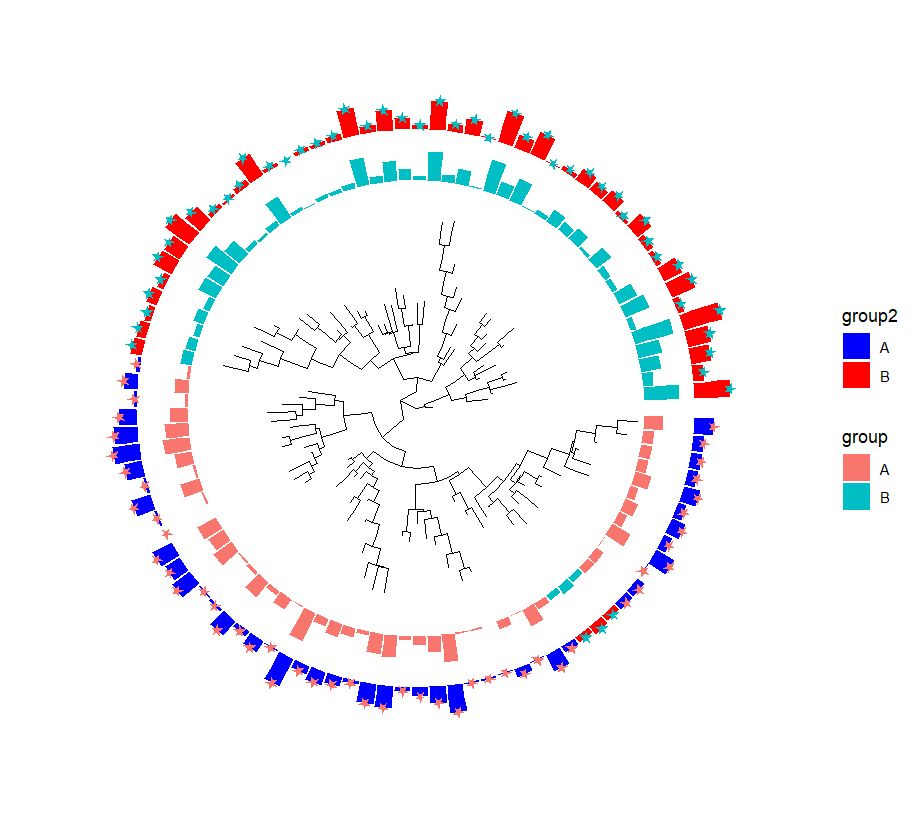
p2 <- p1 +# 使用 geom_fruit_list 函数添加一个水果列表图层,包含两个子图层geom_fruit_list(geom_fruit(data = df,geom = geom_col,mapping = aes(y=id, x=value, fill=group2),), # 第一个子图层,使用 geom_fruit 函数添加柱状图层来显示 df 数据框的数据scale_fill_manual(values=c("blue", "red")), # To map group2new_scale_fill(), # To initialize fill scale.geom_fruit(data = dt,geom = geom_star,mapping = aes(y=id, x=value, fill=group),size = 2.5,color = NA,starstroke = 0) # 第二个子图层,使用 geom_fruit 函数添加星形图层来显示 dt 数据框的水果数据) +new_scale_fill() # 初始化填充颜色比例尺p2
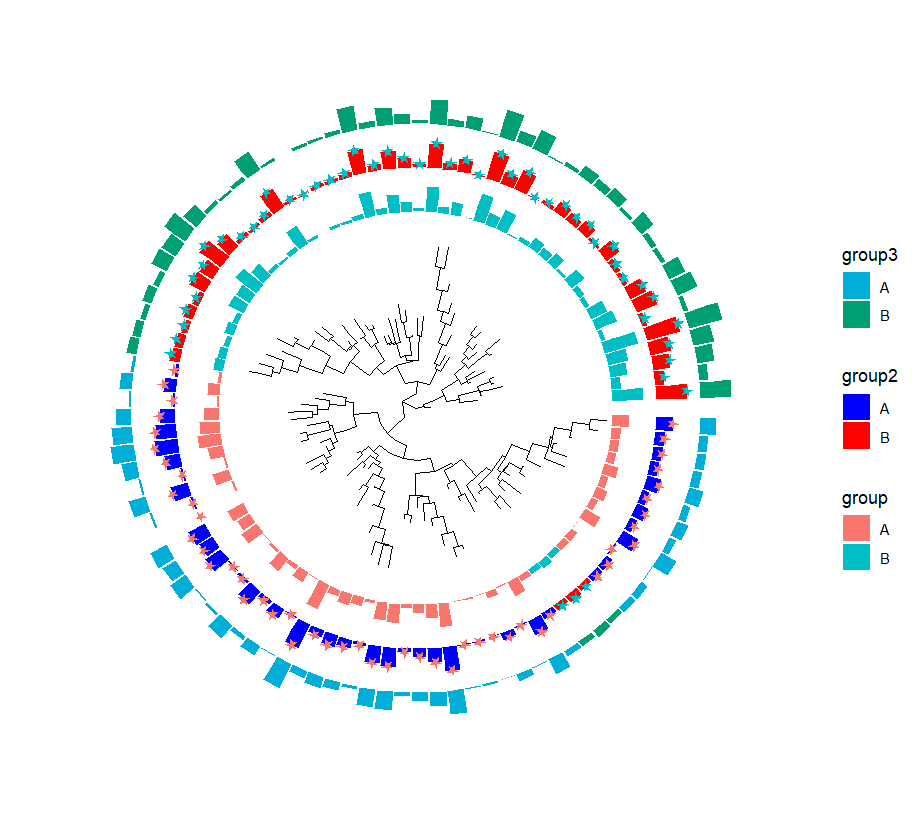
p3 <- p2 +# 添加第三层数据信息geom_fruit(data = dtf,geom = geom_col,mapping = aes(y=id, x=value, fill=group3),stat = "identity") +scale_fill_manual(values=c("#00AED7", "#009E73")) # 设置颜色标尺p3
小果还提供思路设计、定制生信分析、文献思路复现;有需要的小伙伴欢迎直接扫码咨询小果,竭诚为您的科研助力!

定制生信分析
服务器租赁
扫码咨询小果


往期回顾
|
01 |
|
02 |
|
03 |
|
04 |