你不容错过的单细胞分析神器!TSCAN包,预测细胞时间序列,聚类细胞,简直好用到令人难以置信!

公众号后台回复“111”
领取本篇代码、基因集或示例数据等文件
文件编号:240510-1
需要租赁服务器的小伙伴可以扫码添加小果,此外小果还提供生信分析,思路设计,文献复现等,有需要的小伙伴欢迎来撩~

if (!require("BiocManager", quietly = TRUE))install.packages("BiocManager ")BiocManager::install("TSCAN") # 在BiocManager环境下安装TSCAN查看是否安装成功:packageVersion("TSCAN") # 查看TSCAN版本

library(TSCAN) # 载入TSCAN包data(lpsdata) # 载入lpsdata数据class(lpsdata) # 查看lpsdata数据数据类型dim(lpsdata) # 查看lpsdata矩阵的行列信息

procdata <- preprocess(lpsdata) # 对lpsdata数据进行预处理。preprocess函数可以用来处理数据,筛选掉在大多数细胞中不表达或者表达不具有差异性的基因。class(procdata) # 查看procdata数据数据类型dim(procdata) # 查看procdata矩阵的行列信息

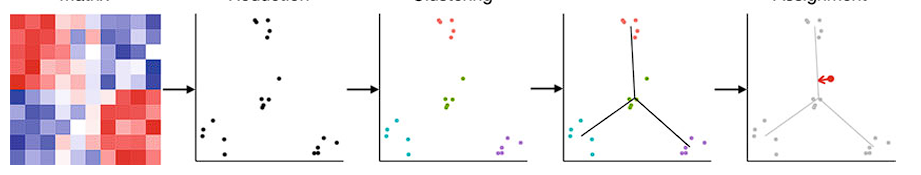
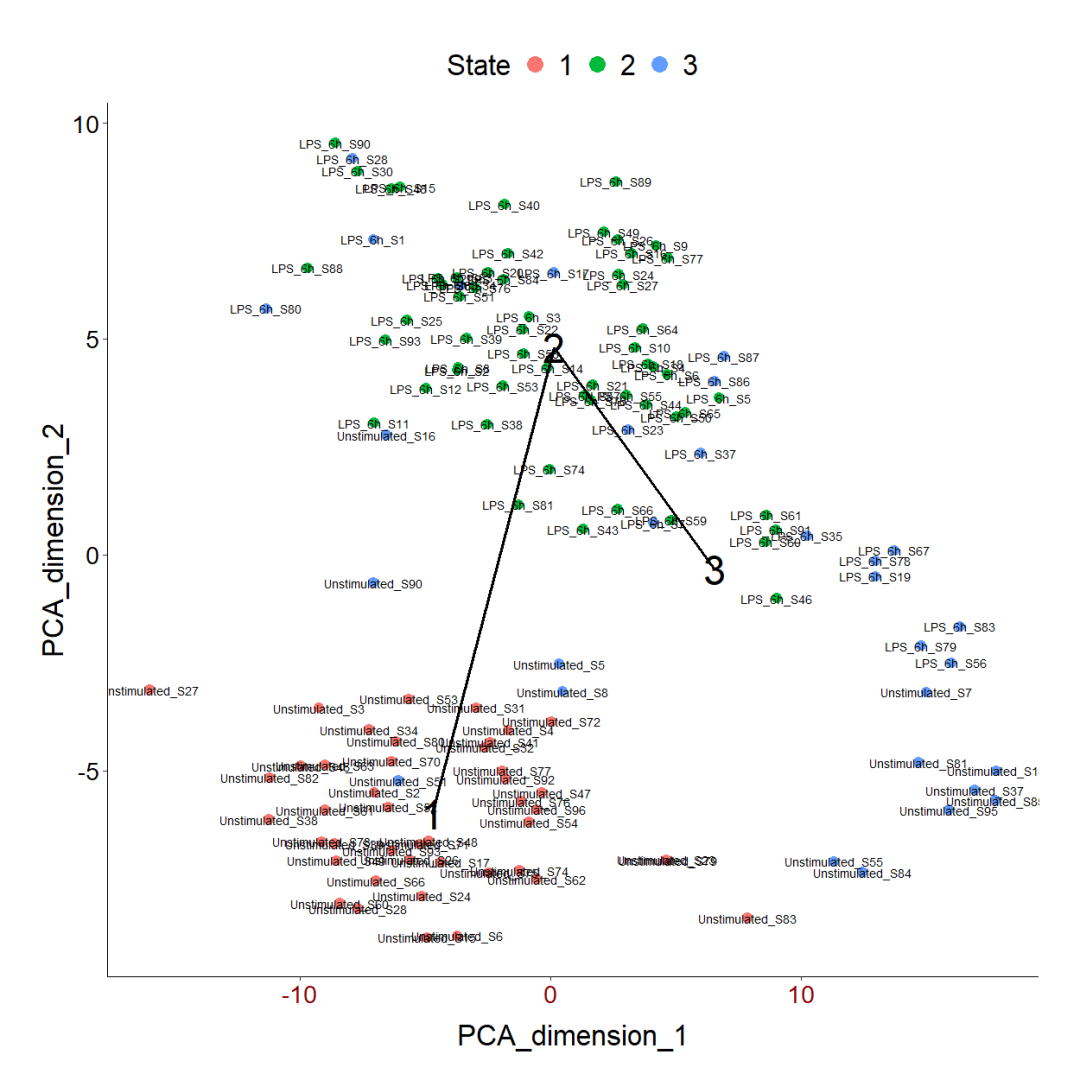
lpsmclust <- exprmclust(procdata) # 使用exprmclust函数对数据进行维度约简接着使用plotmclust函数来对约简之后的数据进行可视化,命令如下:plotmclust(lpsmclust) # 对约简之后的数据进行可视化
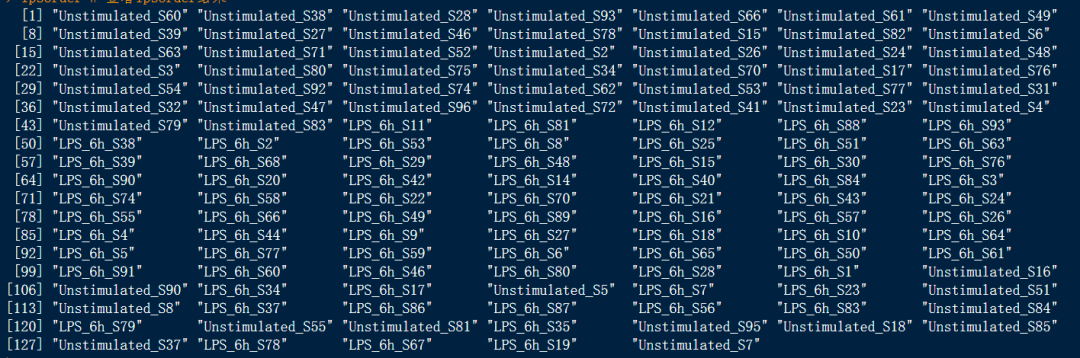
lpsorder <- TSCANorder(lpsmclust) #使用TSCANorder函数来获得TSCAN 的伪时间排序lpsorder # 查看lpsorder结果
diffval <- difftest(procdata,lpsorder) # 使用 difftest 函数来检测在构建的伪时间顺序中差异表达的基因diffval # 显示diffval结果

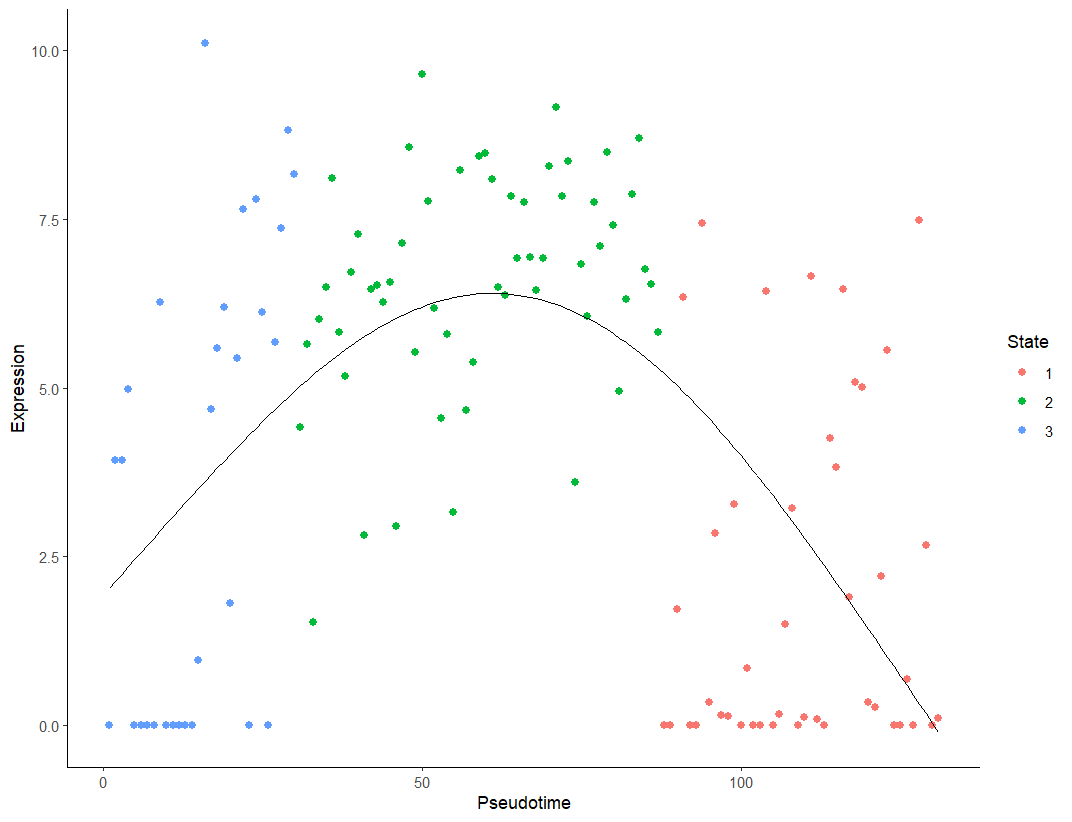
STAT2expr <- log2(lpsdata["STAT2",]+1)singlegeneplot(STAT2expr, TSCANorder(lpsmclust,flip=TRUE,orderonly=FALSE))
小果还提供思路设计、定制生信分析、文献思路复现;有需要的小伙伴欢迎直接扫码咨询小果,竭诚为您的科研助力!

定制生信分析
服务器租赁
扫码咨询小果

往期回顾
|
01 |
|
02 |
|
03 |
|
04 |