前言
谈到单细胞分析,少不了的就是聚类!聚类!在聚类!!不幸的是,这些“以聚类中心”分析方法依赖于准确定义的聚类解决方案来发现有意义的标记特征。由于聚类解决方案在用户定义的聚类分辨率(聚类数)和选择的聚类算法空间内可能有很大的变化范围,这可能导致不一致和不准确的生物学注释,进而影响下游分析的可靠性。这可把小伙伴们愁坏了,费尽九牛二虎之力完成了从降维、聚类到注释,结果却不知道怎么解释了。真是一顿操作猛如虎,一看结果要心梗!为了克服这些局限性,单细胞分析大佬团队提出 SIMBA。对于每个任务,SIMBA 构造一个图,其中不同的实体(即Cell和Feature,即基因或者Peaks)被表示为节点,这些实体之间的关系被编码为边(构建一张图)。一旦图形构建完成,SIMBA 然后应用源自社交网络技术的多关系图嵌入算法以及基于 Softmax 的转换来将节点或实体嵌入到公共的低维空间中,其中细胞和特征可以根据它们的距离进行分析。
SIMBA可以在统一的框架下解决各种单细胞任务,包括:(1)降维;(2)无聚类标记检测;(3)多模态分析;(4)批次校正和组学整合。
有什么生信分析上的问题大家尽管咨询大海哥!没有时间学习的小伙伴们也不要着急哦!有需要生信分析的小伙伴们也可以找大海哥哦!练了十年生信分析的大海哥对于生信分析知识已经如鱼得水从分析到可视化直到你满意为止!
生信数据处理起来占用内存实在太大了,放过自己的电脑吧!大海哥在这里给大家送上福利了,有需要服务器的小伙伴们,欢迎大家联系大海哥,保证服务器的性价比最高哦
代码教程
在本教程中,我们将利用 10X PBMC scRNA-seq 数据集来展示在 scRNA-seq 数据集上运行 SIMBA 的步骤。这里的数据可以替换为你自己需要分析的数据集,只要保证输入数据的格式为anndata格式即可,这里我们使用的是pbmc3k数据集,这也是一个非常经典的公共数据集。输入数据10多个G,下载链接私信大海哥获取哦!
1. 安装SIMBA包及导入相关依赖包
对于首次使用 conda 的用户,请使用以下命令执行 Bioconda 的一次性设置:
conda config –add channels defaults
conda config –add channels bioconda
conda config –add channels conda-forge
conda config –set channel_priority strict
要使用 conda 安装 simba,请运行:
conda install -c bioconda simba
建议:在新的虚拟环境中安装 simba:
conda create -n env_simba simba
conda activate env_simba
要在 GitHub 上安装最新版本,请执行以下操作:
首先安装simba_pbg
conda install -c bioconda simba_pbg
然后运行:
git clone https://github.com/huidongchen/simba.git
pip install simba –user
或
pip install git+https://github.com/huidongchen/simba
import os
import simba as si
si.__version__
workdir = ‘result_simba_rnaseq’
si.settings.set_workdir(workdir)
si.settings.set_figure_params(dpi=80,
style=’white’,
fig_size=[5,5],
rc={‘image.cmap’: ‘viridis’})
# make plots prettier
from matplotlib_inline.backend_inline import set_matplotlib_formats
set_matplotlib_formats(‘retina’)
2. 加载示例数据并进行预处理
我们读取测试数据,随后我们对测试数据集进行质控和过滤,以滤除表达量较低的基因,这样可以减小数据维度,降低数据稀疏性并提高下游分析的速度和准确性。
adata_CG = si.datasets.rna_10xpmbc3k()
adata_CG
si.pp.filter_genes(adata_CG,min_n_cells=3)
si.pp.cal_qc_rna(adata_CG)
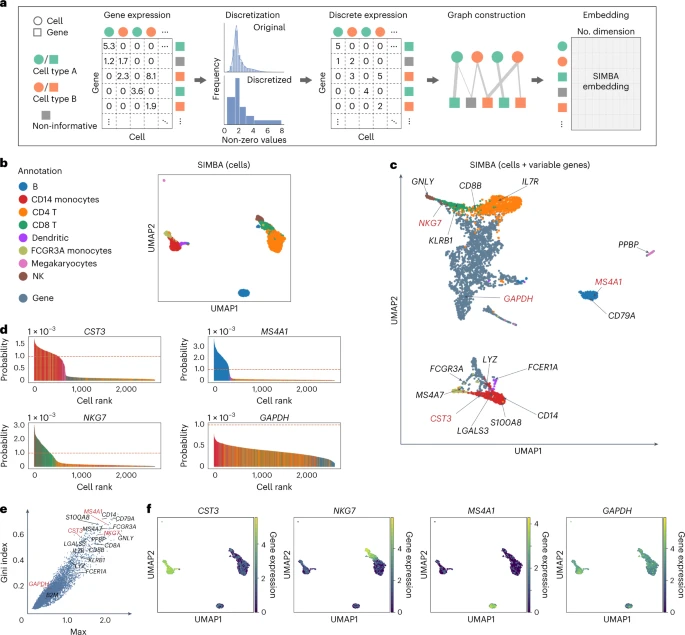
si.pl.violin(adata_CG,list_obs=[‘n_counts’,’n_genes’,’pct_mt’])
我们根据人为设定的阈值过滤低质量细胞,并对计数矩阵的表达值做标准化和对数化处理,然后计算前2000个高变基因并对其可视化。
请注意,是否计算高变基因对图嵌入的结果并没有本质影响,但是计算高变基因可以降低模型的计算时间,与之相对于,如果仅使用高变基因进行图嵌入计算,最终生成的低维表示图中也只包含高变基因。
si.pp.filter_cells_rna(adata,min_n_genes=100)
si.pp.normalize(adata_CG,method=’lib_size’)
si.pp.log_transform(adata_CG)
也可以执行可变基因选择步骤,也可以不执行,不影响R包的计算结果。
si.pp.filter_cells_rna(adata,min_n_genes=100)
si.pp.normalize(adata_CG,method=’lib_size’)
si.pp.log_transform(adata_CG)
3. 离散化 RNA 表达
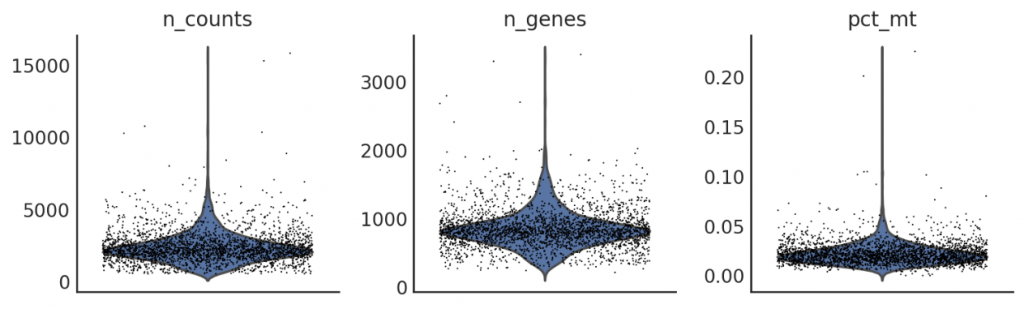
此步骤将非零基因表达值分组为不同的水平,同时保留原始分布,从而在随后的训练中识别不同水平的基因表达。离散化的表达值将用于构建基因-细胞图中的基因-细胞边的权重。
si.tl.discretize(adata_CG,n_bins=5)
si.pl.discretize(adata_CG,kde=False)
4. 生成用于训练的图形
每个 Anndata 对象中的观测值和变量都表示为节点。list_CG中指定的anndata对象为输入编码数据,adata.var_names和data.obs_names将被编码为节点,对于基因表达数据,在默认情况下,不同水平的基因表达是由不同权重的节点-节点之间边编码的。
si.tl.gen_graph(list_CG=[adata_CG],
layer=’simba’,
use_highly_variable=False,
dirname=’graph0′)
5.模型训练
接下来我们可以训练模型了。作者给出了修改模型训练参数的代码,但是在一般情况下,我们使用默认参数就可以很好的训练模型。通常,唯一可能需要调整的参数是权重衰减wd。在大多数情况下,自动确定的值(通过启用实现)可以充分执行:auto_wd=True。如果不需要对参数进行调整,则只需使用以下方法即可执行训练:
si.tl.pbg_train(auto_wd=True, save_wd=True, output=’model’)
在这里,我们演示了如何在必要时修改与训练相关的参数。
# 更改参数
dict_config = si.settings.pbg_params.copy()
# dict_config[‘wd’] = 0.015521
dict_config[‘wd_interval’] = 10 # we usually set `wd_interval` to 10 for scRNA-seq datasets for a slower but finer training
dict_config[‘workers’] = 12 #The number of CPUs.
## 开始训练
si.tl.pbg_train(pbg_params = dict_config, auto_wd=True, save_wd=True, output=’model’)
我们也可以可视化出模型训练指标的变化情况。通过绘制训练指标以监控过度拟合并评估最终模型。理想情况下,经过一定数量的训练后,度量曲线应该保持稳定。默认情况下,这行代码会显示了训练损失(越低越好)以及验证损失(越低越好)和 MRR (越高越好)。
si.pl.pbg_metrics(fig_ncol=1)
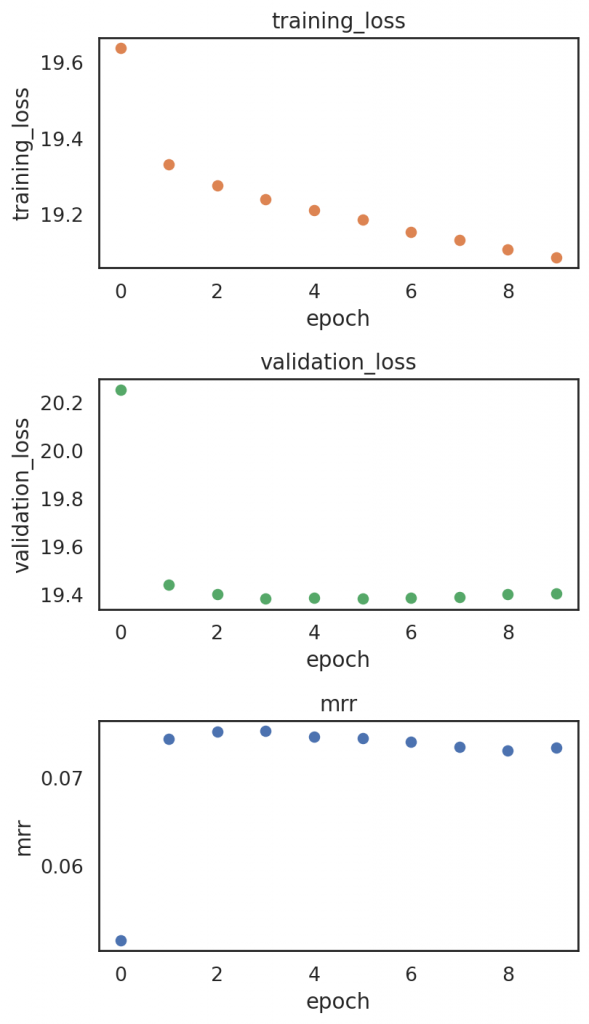
如果需要进一步的参数调整,可以通过更新变量来实现在同一图上训练时使用更新的参数生成新的嵌入,例如,output
si.tl.pbg_train(pbg_params = dict_config, auto_wd=True, save_wd=True, output=”model2″)
如果需要,一旦训练参数最终确定,可以使用以下函数更新 PBG 训练参数:
si.settings.set_pbg_params(dict_config)
要加载训练的结果,可以执行以下步骤:默认情况下,它使用存储在.setting.pbg_params
# load back ‘graph0’
si.load_graph_stats()
# load back ‘model’ that was trained on ‘graph0’
si.load_pbg_config()
用户还可以指定不同的路径,例如,
# load back ‘graph1’
si.load_graph_stats(path=’./result_simba_rnaseq/pbg/graph1/’)
# load back ‘model2’ that was trained on ‘graph1’
si.load_pbg_config(path=’./result_simba_rnaseq/pbg/graph1/model2/’)
6. 训练后分析
# 读取从从PBG培训获得的实体嵌入。
dict_adata = si.read_embedding()
dict_adata
adata_C = dict_adata[‘C’] #细胞嵌入
adata_G = dict_adata[‘G’] # 基因嵌入
7. 可视化细胞和基因的嵌入
接下来我们首先可视化出细胞的Umap图,基于这50维低维表示,我们可以将其作为umap计算的输入,并且得到其Umap坐标,并可视化。
##添加细胞类型注释
adata_C.obs[‘celltype’] = adata_CG[adata_C.obs_names,:].obs[‘celltype’].copy()
adata_
palette_celltype={‘B’:’#1f77b4′,
‘CD4 T’:’#ff7f0e’,
‘CD8 T’:’#279e68′,
‘Dendritic’:”#aa40fc”,
‘CD14 Monocytes’:’#d62728′,
‘FCGR3A Monocytes’:’#b5bd61′,
‘Megakaryocytes’:’#e377c2′,
‘NK’:’#8c564b’}
si.tl.umap(adata_C,n_neighbors=15,n_components=2)
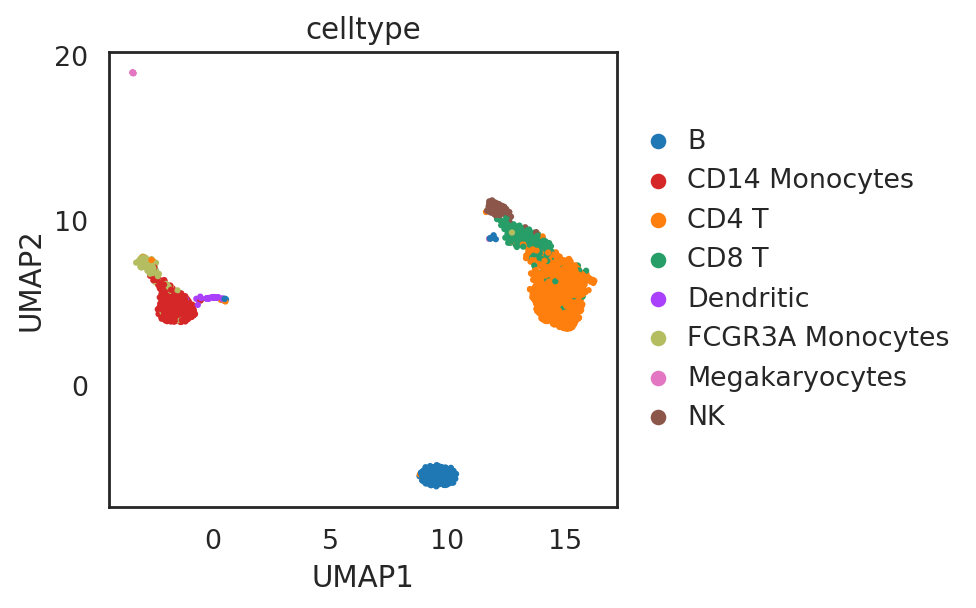
si.pl.umap(adata_C,color=[‘celltype’],
dict_palette={‘celltype’: palette_celltype},
fig_size=(6,4),
drawing_order=’random’)
然后我们可以计算并得到细胞和基因的联合嵌入,并将其可视化在Umap图上。
# 将细胞和基因嵌入同一空间
adata_all = si.tl.embed(adata_ref=adata_C,list_adata_query=[adata_G])
adata_all.obs.head()
## 添加细胞和基因注释
adata_all.obs[‘entity_anno’] = “”
adata_all.obs.loc[adata_C.obs_names, ‘entity_anno’] = adata_all.obs.loc[adata_C.obs_names, ‘celltype’]
adata_all.obs.loc[adata_G.obs_names, ‘entity_anno’] = ‘gene’
adata_all.obs.head()
palette_entity_anno = palette_celltype.copy()
palette_entity_anno[‘gene’] = “#607e95”
si.tl.umap(adata_all,n_neighbors=15,n_components=2)
si.pl.umap(adata_all,color=[‘id_dataset’,’entity_anno’],
dict_palette={‘entity_anno’: palette_entity_anno},
drawing_order=’original’,
fig_size=(6,5))

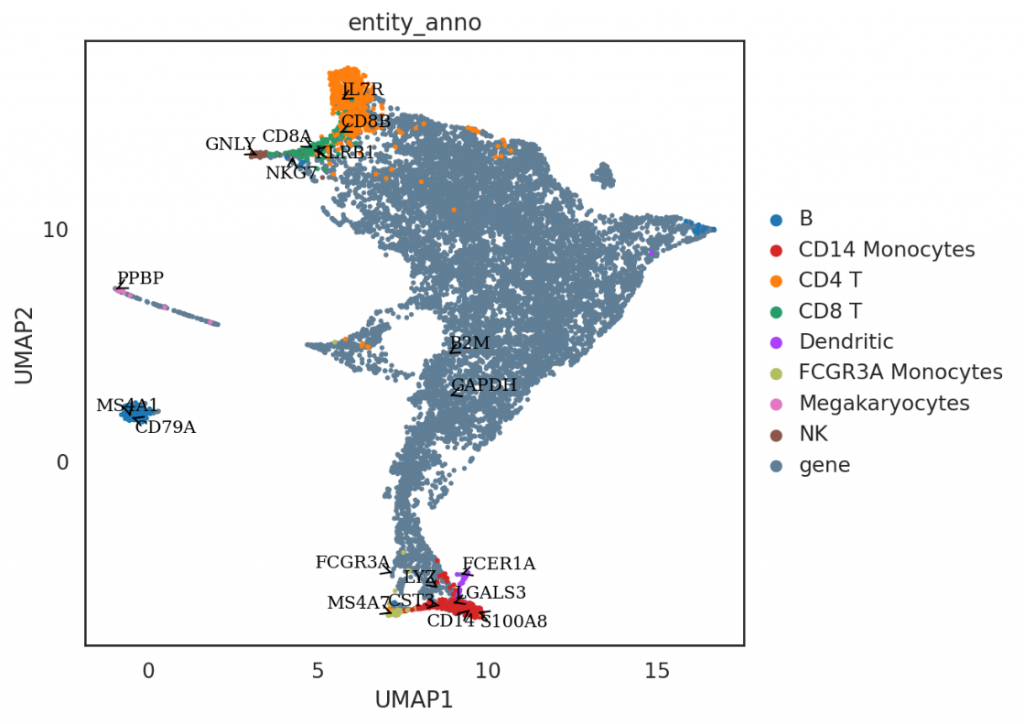
作者同样提供了工具,帮助我们可视化出不同基因在Umap图上的位置,并且给出文字表示。
图中除了GAPDH基因和B2M基因是管家基因,其他基因都是特定细胞亚群的标记基因。在Umap图上,实际上这些Marker基因的Umap坐标都处于对应的亚群内,而管家基因的坐标则不具有亚群特异性。
#在共嵌入空间中突出一些标记基因
marker_genes = [‘IL7R’, ‘CD79A’, ‘MS4A1’, ‘CD8A’, ‘CD8B’, ‘LYZ’, ‘CD14’,
‘LGALS3’, ‘S100A8’, ‘GNLY’, ‘NKG7’, ‘KLRB1’,
‘FCGR3A’, ‘MS4A7’, ‘FCER1A’, ‘CST3’, ‘PPBP’]
si.pl.umap(adata_all[::-1,],color=[‘entity_anno’],dict_palette={‘entity_anno’: palette_entity_anno},
drawing_order=’original’,
texts=marker_genes + [‘GAPDH’, ‘B2M’],
show_texts=True,
fig_size=(8,6))
我们还可以按照以下步骤仅可视化部分基因(例如,可变基因)而不是所有基因:
# 获取可变基因
si.pp.select_variable_genes(adata_CG, n_top_genes=3000)
var_genes = adata_CG.var_names[adata_CG.var[‘highly_variable’]].tolist()
# 获得细胞和可变基因的SIMBA包埋
adata_all2 = adata_all[list(adata_C.obs_names) + var_genes,].copy()
# 使用UMAP可视化
si.tl.umap(adata_all2,n_neighbors=15,n_components=2)
si.pl.umap(adata_all2,color=[‘entity_anno’],
dict_palette={‘entity_anno’: palette_entity_anno},
drawing_order=’original’,
fig_size=(6,5))
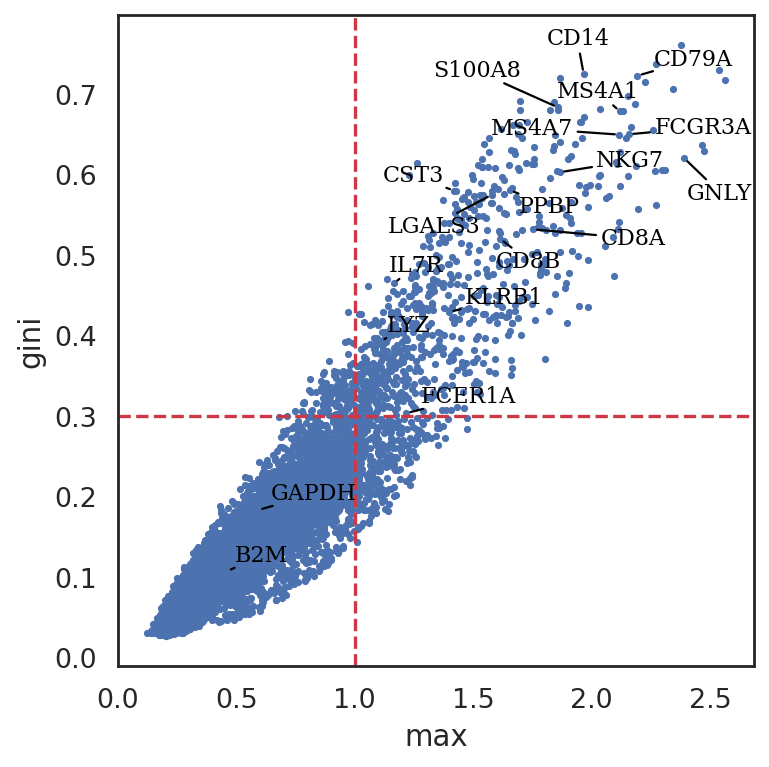
计算SIMBA指标,包括最大值、基尼指数、标准差和熵,以评估基因的细胞类型特异性:max:值越高表示细胞类型特异性越高
基尼系数:值越高表示细胞类型特异性越高
STD:值越高表示细胞类型特异性越高
熵:值越低表示细胞类型特异性越高
adata_cmp = si.tl.compare_entities(adata_ref=adata_C, adata_query=adata_G)
adata_cmp
# 基因可以根据一个或多个指标进行排名
adata_cmp.var.head()
# SIMBA指标可以使用以下函数进行可视化:
si.pl.entity_metrics(adata_cmp,
x=’max’,
y=’gini’,
show_contour=False,
texts=marker_genes + [‘GAPDH’, ‘B2M’],
show_texts=True,
show_cutoff=True,
size=5,
text_expand=(1.3,1.5),
cutoff_x=1.,
cutoff_y=0.3,
save_fig=False)
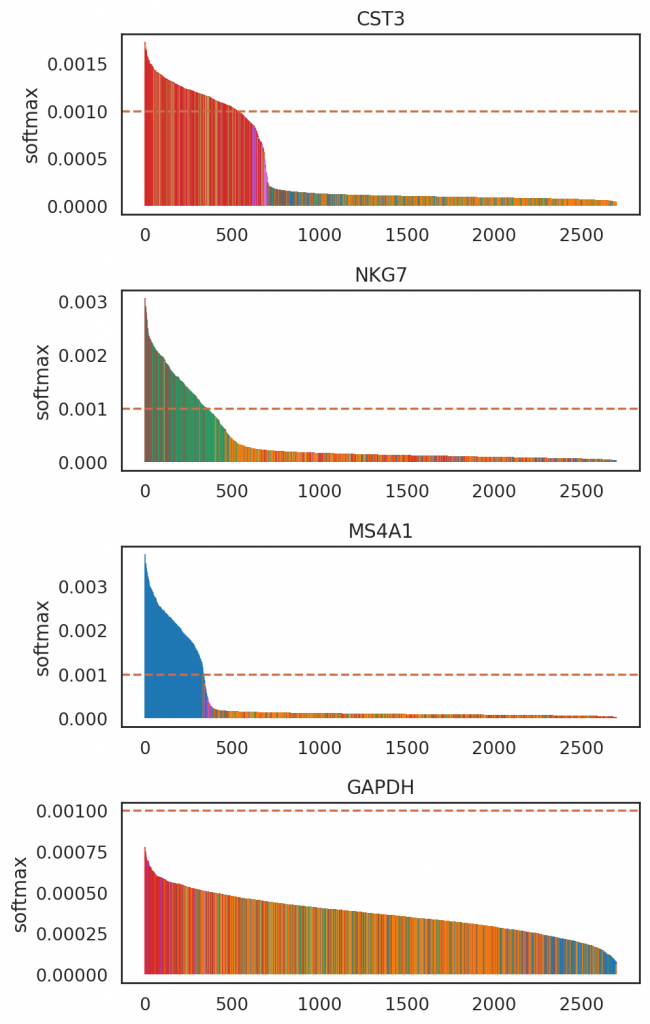
使用SIMBA 条形码图基于边缘置信度可视化特征细胞概率:-不平衡表示细胞类型特异性基因-均匀分布表示非特异性基因。
# 添加细胞注释
adata_cmp.obs[‘celltype’] = adata_CG.obs.loc[adata_cmp.obs_names,’celltype’]
list_genes = [‘CST3’, ‘NKG7’, ‘MS4A1’, ‘GAPDH’]
si.pl.entity_barcode(adata_cmp,
layer=’softmax’,
entities=list_genes,
anno_ref=’celltype’,
show_cutoff=True,
cutoff=0.001,
palette=palette_celltype,
fig_size=(6, 2.5),
save_fig=False)
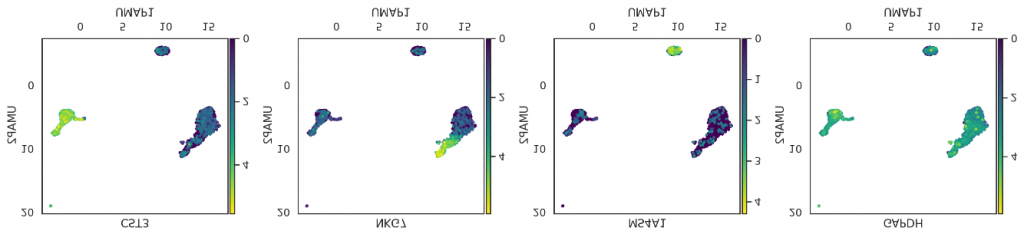
同样的基因列表也可以在UMAP上可视化,以确认它们的细胞类型特异性
adata_CG.obsm[‘X_umap’] = adata_C[adata_CG.obs_names,].obsm[‘X_umap’].copy()
si.pl.umap(adata_CG,
color=[‘CST3’, ‘NKG7’, ‘MS4A1’, ‘GAPDH’],
drawing_order=’sorted’,
size=5,
alpha=0.9,
fig_ncol=4,
fig_size=(4,4),
save_fig=False)
小结
SIMBA的优点是可以整合不同方法得到的基因,比如细胞通讯、MOFA(多组学因子分析,一种多组学分析算法)的feature、GLUE的GRN(高歌老师开发的scRNA-seq+scATAC-seq配对算法生成的转录因子调控网络)、pySCENIC生成的调节子活性矩阵、还有细胞的marker,可以在embedding层面,研究他们的相关性和特征。此外,SIMBA在scATAC-seq分析,scRNA+scATAC-seq整合分析的层面,可能会有更好的效果。最后大海哥给大家介绍一个云工具!同学们如果觉得自己的代码水平一般,对于很多的参数不知道怎么改,可以体验一下我们的云生信小工具,只需输入数据,即可轻松生成所需图表,字体大小、标题等也可一键更改。感兴趣的小伙伴去云生信(http://www.biocloudservice.com/home.html)体验一下吧!
